Всем привет! Недавно я закончил один из этапов собственного проекта, в котором я провел сравнительный анализ 3 одних из самых известных нейросетей для семантической сегментации: U-Net, LinkNet, PSPNet. Теперь я хочу поделиться со всеми, чтобы в случае, если кто-то захочет сделать что-то подобное или ему просто понадобится, то он не искал весь интернет, как я, а легко и просто все нашел. В конце главы каждый нейросети я оставил ссылки на оригинальные статьи для желающих самостоятельно все изучить (на английском). Ссылка на мой GitHub с полноценной версией всех нейросетей и main файла в конце статьи.
Я расскажу кратко о подготовке входных данных перед тем, как подавать их в нейросеть, а также объясню самые важные детали каждой модели по отдельности. Использовал я библиотеку Tensorflow, а обучение проводил в среде Google Colab.
Семантическая сегментация
Семантическая сегментация - это метод компьютерного зрения, который позволяет не только обнаруживать объекты на изображениях, но и определяет их точное пространственное местоположение, классифицируя каждый пиксель. Эта техника полезна, когда изображение
необходимо разделить на несколько категорий (многоклассовая классификация), а не просто провести различие между двумя классами (бинарная классификация). Она относится к Supervised Learning, то есть помимо самих изображений, нужны и их идеальные данные (ground truth), на которых она будет опираться во время предсказаний.
Подготовка данных
Прежде, чем отдавать изображения на вход нейросети, необходимо их преобразовать в нужный формат и оптимизировать.
Сначала мы загружаем изображения и маски с помощью OpenCV и сразу через функцию imread() преобразуем из RGB в диапазон серого (grayscale). Таким образом мы облегчаем данные для нейросети, что упрощает обучение, но при этом эффективность остается той же. После с помощью array() маски и изображения мы представляем данные в виде массивов.
BASE_PATH = Path("/content/drive/MyDrive/Colab Notebooks/")
IMAGE_DIR = BASE_PATH / "images-3"
MASK_DIR = BASE_PATH / "masks-3"
train_images = []
for img_path in sorted(IMAGE_DIR.glob("*.jpg")):
img = cv2.imread(str(img_path), 0)
img = cv2.resize(img, (SIZE_Y, SIZE_X))
train_images.append(img)
train_images = np.array(train_images)
train_masks = []
for mask_path in sorted (MASK_DIR.glob("*.png")):
mask = cv2.imread(str(mask_path), 0)
mask = cv2.resize(mask, (SIZE_Y, SIZE_X), interpolation = cv2.INTER_NEAREST)
train_masks.append(mask)
train_masks = np.array(train_masks)Далее нам необходимо упорядочить классы масок в порядке возрастания, начиная от 0. Используется только в случае, когда классов несколько. Мы сохраняем из 3-х мерного массива train_masks три значения (n - классы, h-высота, w- ширина), чтобы после сортировки и упорядочивания вернуть в исходные размеры. Для того чтобы применить LabelEncoder, который работает с одномерными последовательностями, весь массив масок разворачивается в форму (n*h*w, 1) с помощью reshape(). fit_transformer() находит все уникальные значения (то есть классы) в масках и сопоставляет их с целыми числами, начиная с 0. Это необходимо, чтобы привести метки к последовательному виду (например, 0, 1, 2, …), что важно для вычисления функции потерь, такой как функция потери Categorical Crossentropy ожидает, что классы будут представлены именно в таком виде. После кодирования одномерный массив возвращается в исходную размерность (n, h, w), но уже с обновлёнными, упорядоченными метками.
from sklearn.preprocessing import LabelEncoder
labelencoder = LabelEncoder()
n, h, w = train_masks.shape
train_masks_reshaped = train_masks.reshape(-1,1)
train_masks_reshaped_encoded = labelencoder.fit_transform(train_masks_reshaped.ravel()) 0
train_masks_encoded_original_shape = train_masks_reshaped_encoded.reshape(n, h, w) Исходный массив данных является 3-х мерным, но сверточные нейросети ожидают входной 4D тензор в форме (n, h, w, channels). Так как изображения и маски в градациях серого, количество каналов равно 1. Функция np.expand_dims(....., axis=3) добавляет новую ось, превращая форму в (n, h, w, 1). Для улучшения сходимости модели во время обучения мы нормализует значения пикселей, переводя их из диапазона [0, 255] в диапазон [0, 1].
train_images = np.expand_dims(train_images, axis=3)
train_images = train_images / 255.0
train_masks_input = np.expand_dims(train_masks_encoded_original_shape, axis=3) usedИ наконец мы преобразуем маски с целочисленными значениями в формат One-hot encoding. Теперь каждая метка преобразуется в вектор длиной n_classes, где на позиции истинного класса стоит 1, а на остальных – 0. Такой формат требуется для вычисления функции потерь categorical crossentropy.
y_train_cat = to_categorical(y_train, num_classes=n_classes)
y_val_cat = to_categorical(y_val, num_classes=n_classes)
y_test_cat = to_categorical(y_test, num_classes=n_classes)Поздравляю! Мы завершили основную подготовку входных данных. Это не весь код, но самое главное, по-моему, не упустил. Переходим теперь к нейросетям.
U-Net
U-Net- нейросеть кодер-декодер архитектуры представленный в 2015 году для сегментации медицинских снимков. Благодаря своей высокой точности и способности работать с малыми датасетами, U-Net стал одной из самых популярных нейросетей для сематической сегментации (рис. 1).
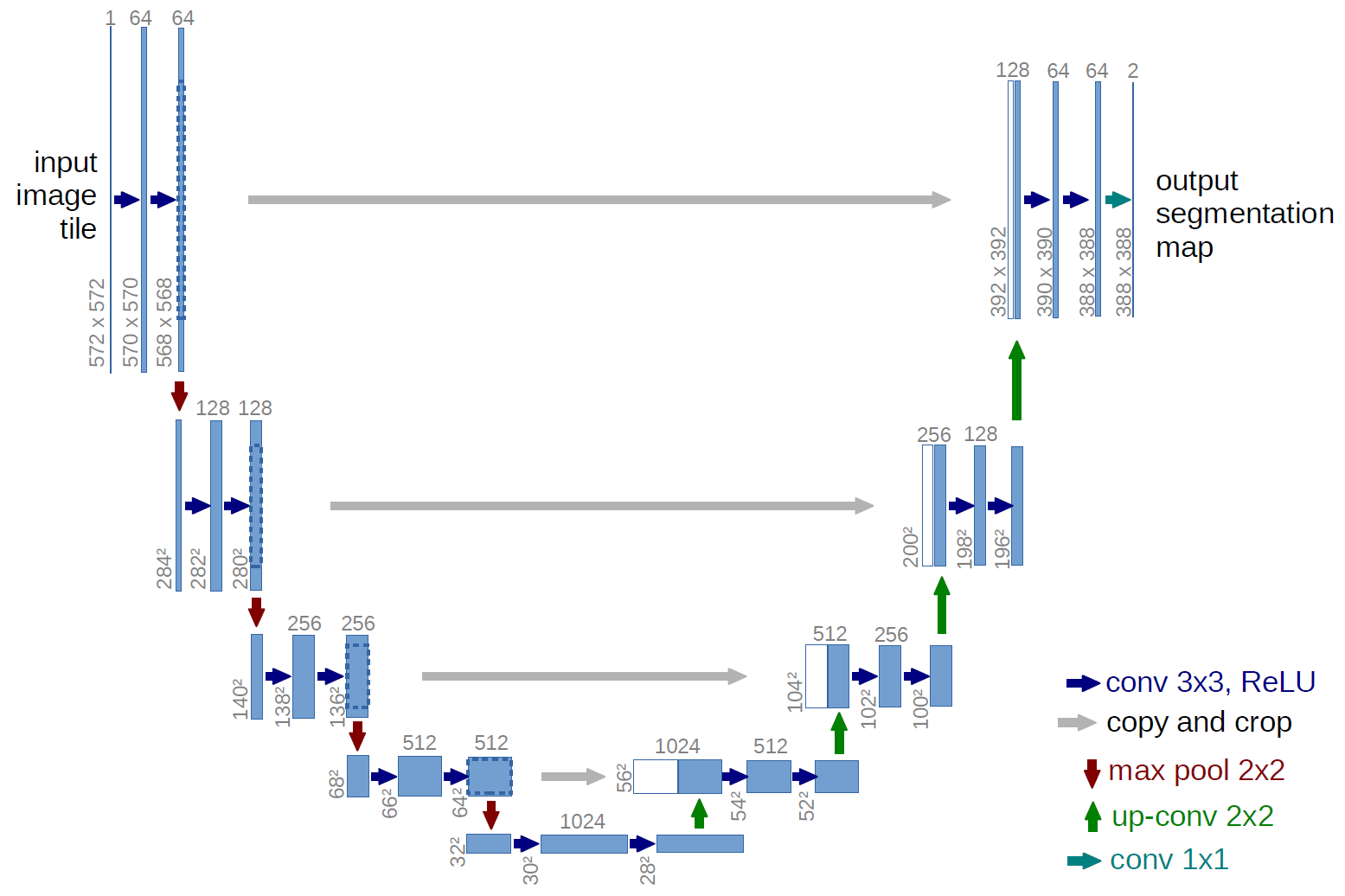
Кодер состоит из четырех идентичных блоков, в каждом из которых последовательно применяются два сверточных преобразования Conv2D() с ядрами 3х3 для извлечения все более детальных признаков, после чего выполняется операция Max Pooling 2х2 (красная стрелка) для уменьшения размера карты признаков вдвое. Перед пулингом мы сохраняем признаки в пропускных соединениях (skip_connection), которые потом передадим в декодер блока. С каждым блоком количество фильтров input_filters, которые представляют количество карт признаков, увеличивается. Стандартом является 64-128-256-512, но в своих экспериментах я применил облегченную версию с фильтрами 16-32-64-128. Она была менее ресурсоемкой, но при этом довольно эффективной.
def encoder_block(x, input_filters: int):
x = Conv2D(input_filters, (3, 3), kernel_initializer='he_normal', padding='same', use_bias=False)(x)
x = BatchNormalization()(x)
x = ReLU()(x)
x = Dropout(0.1)(x)
x = Conv2D(input_filters, (3, 3), kernel_initializer='he_normal', padding='same', use_bias=False)(x)
x = BatchNormalization()(x)
x = ReLU()(x)
skip_connection = x
x = MaxPooling2D((2, 2))(x)
return x, skip_connectionЗатем все полученные карты признаков передаются уже декодеру через bottleneck - самую глубокую часть сети, где происходит максимальное сжатие информации перед ее восстановлением в декодере. Он состоит из двух сверточных слоев Conv2D(). Полностью идентичен кодер блоку, но отсутствует skip_connection и Max Pooling(). Входные фильтры в количестве 1024 или 256 в облегченной версии.
def decoder_block(x, skip_connection, input_filters: int):
x = Conv2DTranspose(input_filters, (2, 2), strides=(2, 2), padding='same', use_bias=False)(x)
x = BatchNormalization()(x)
x = ReLU()(x)
x = concatenate([x, skip_connection])
x = Conv2D(input_filters, (3, 3), kernel_initializer='he_normal', padding='same', use_bias=False)(x)
x = BatchNormalization()(x)
x = ReLU()(x)
x = Dropout(0.2)(x)
x = Conv2D(input_filters, (3, 3), kernel_initializer='he_normal', padding='same', use_bias=False)(x)
x = BatchNormalization()(x)
x = ReLU()(x)
return xДекодер состоит из блоков, аналогичных блокам кодера, но вместо Max Pooling используется транспонированная свертка Conv2DTranspose (зеленая стрелка), которая благодаря шагу (strides=(2, 2)) удваивает пространственный размер карты признаков. На выходе U-сети формируется семантическая маска, соответствующая размерам исходного изображения.
Важно отметить, что Dropout() в каждом блоке разный. Он случайным образом отключает долю нейронов на каждом шаге обучения, тем самым уменьшая риск переобучения. В кодере модель извлекает базовые признаки и он менее склонен к переобучению, поэтому 0.1, а bottleneck, наоборот, уже работает с высокоуровневые и абстрактными признаки, также здесь содержится множество параметров, что повышает риски переобучения. Декодер работает как с низкоуровневыми, так и с высокоуровневыми признаками, а потому здесь важен баланс. Оригинальная статья: U-Net: Convolutional Networks for Biomedical Image Segmentation
LinkNet
LinkNet была представлена в 2017 году как модель семантической сегментации, предназначенная для обработки изображений в режиме реального времени (рис. 2). Основная идея заключается в эффективной передаче извлеченных признаков между слоями кодера и декодера слоями с помощью пропускных соединений, что повышает точность и снижает потери пространственной информации.
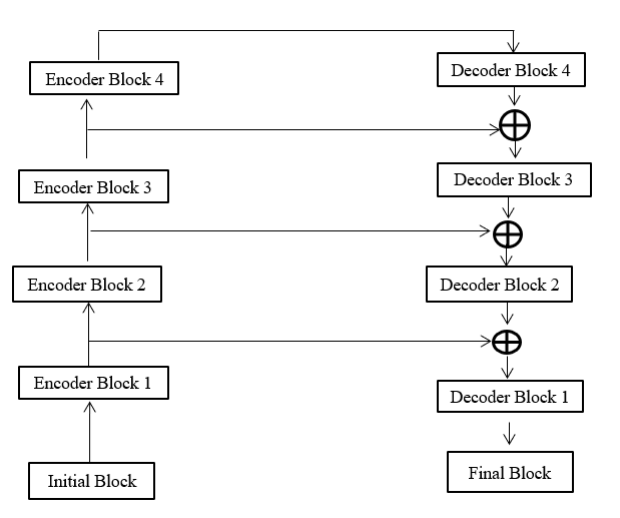
LinkNet начинается с начального блока, который применяет свертку входного изображения с использованием ядра размером 7x7 и шагом 2x2, а затем выполняет Max-pooling в области 3x3 и шагом 2x2.
initial = Conv2D(64, kernel_size=(7,7), strides=2, use_bias = False, padding='same', kernel_initializer='he_normal')(inputs)
initial = Dropout(0.1)(initial)
initial = BatchNormalization()(initial)
initial = ReLU()(initial)
initial = MaxPooling2D((3,3), strides=2, padding='same')(initial)
Затем идут 4 кодер блока (рис. 3), каждый из которых состоит из двух остаточных блоков и пропускных соединений по принципу нейросети классификатора ResNet. Единственным отличием является разница в том, что шаг /2 используется в первых же остаточных блоках, а не после двух первых, как в ResNet.
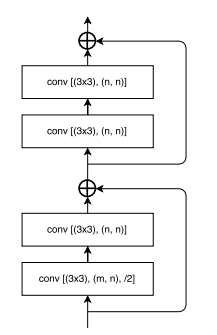
Стрелками обозначены пропускные связи (skip connection), а перекрестье в круге- это функция add(), которая просто складывает воедино карты признаков, которые прошли через две свертки с картами признаков, которые были сохранены с помощью skip connection перед Conv2D. В первом остаточном блоке обязательно необходимо провести данные через сверточный слой ядром 1х1 и шагом /2, чтобы оно соответствовало шагу первого остаточного блока и следовательно избежать ошибок. Буквами m и n на рис.3 представлены количество входных и выходных карт признаков, начиная с 64. В первом кодере блоке m и n идентичны, когда в 3 последующих n всегда в два раза больше m, то есть 64&128, 128&256 и 256&512.
shortcut_1 = Conv2D(input_filters, kernel_size=(1, 1), strides=2, use_bias=False, padding='same', kernel_initializer='he_normal')(x)
shortcut_1 = BatchNormalization()(shortcut_1)
x = Conv2D(input_filters, kernel_size=(3, 3), strides=2, use_bias=False, padding='same', kernel_initializer='he_normal')(x)
x = BatchNormalization()(x)
x = ReLU()(x)
x = Conv2D(input_filters, kernel_size=(3, 3), use_bias=False, padding='same', kernel_initializer='he_normal')(x)
x = BatchNormalization()(x)
x = ReLU()(x)
x = add([x, shortcut_1])
Далее переходим к декодер блоку (рис.4). Их также всего 4 блока, но его строение значительно проще. Состоит из двух сверточных слоев с ядрами 1х1 и транспонированной сверткой с ядром 3х3, задача которого состоит в увеличении размера карт признаков в 2 раза с помощью шага *2. С каждым уровнем декодера количество каналов карты признаков также уменьшается в два раза начиная с 512.
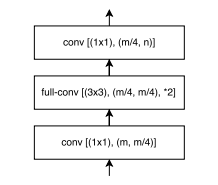
Можно заметить, что входные и выходные карты признаков представлены одной буквой m. Однако в первом сверточном слое их сокращают в 4 раза. В оригинальной статье не упоминается с какой целью это было сделано, но можно предположить с целью оптимизации данных перед тем, как подавать их в транспонированную свертку. В последнем сверточном слое количество выходных карт признаков n в два раза меньше входного m.
def decoder_block(x, skip_connection, input_filters, output_filters: int):
x = Conv2D(input_filters // 4, kernel_size=(1, 1), use_bias = False, padding='same', kernel_initializer='he_normal')(x)
x = BatchNormalization()(x)
x = ReLU()(x)
x = Conv2DTranspose(input_filters // 4, kernel_size=(3,3), strides=2, use_bias = False, padding='same', kernel_initializer='he_normal')(x)
x = BatchNormalization()(x)
x = ReLU()(x)
x = Dropout(0.1)(x)
x = Conv2D(output_filters, kernel_size=(1, 1), use_bias = False, padding='same', kernel_initializer='he_normal')(x)
x = BatchNormalization()(x)
if skip_connection is not None:
x = add([x, skip_connection])
x = ReLU()(x)
return x
Если еще раз посмотреть на архитектуру LinkNet (рис.2), то можно заметить, что там также применяется функция сложения add() для признаков предыдущего декодер блока и признаки, переданные с помощью skip connection от кодера блока того же уровня, что и следующий декодер блок. Это все необходимо, как и в U-Net, для восстановления пространственной информации и создания точной сегментационной маски на выходе нейросети. Проверка пропускного соединения на None необходима, так как всего функций сложения будет только 3, и для последнего декодера нам не нужно сохранять переменную skip_connection.
Последним этапом является финальный блок, состоящий из двух транспонированных сверток и одной обычной свертки. Количество входных признаков у последней транспонированной свертки равно количеству классов вашего датасета. Сам же финальный блок обеспечивает полное восстановление пространственной детализации и формирует финальную предсказательную карту, которая затем нормируется функцией Softmax, если у вас задача многоклассовой сегментации, для получения окончательного решения по каждому пикселю. Если у вас бинарная классификация, то Softmax необходимо заменить на сигмоидальную функцию. Оригинальная статья: LinkNet: Exploiting Encoder Representations for Efficient Semantic Segmentation
out = Conv2DTranspose(32, kernel_size=(3,3), strides=2, use_bias = False, padding='same', kernel_initializer='he_normal')(d1)
out = BatchNormalization()(out)
out = ReLU()(out)
out = Conv2D(32, kernel_size=(3,3), use_bias = False, padding='same', kernel_initializer='he_normal')(out)
out = BatchNormalization()(out)
out = ReLU()(out)
out = Conv2DTranspose(n_classes, kernel_size=(2,2), strides=2, use_bias = False, padding='same', kernel_initializer='he_normal')(out)
out = BatchNormalization()(out)
outputs = Softmax()(out)PSPNet
Модель семантической сегментации Pyramid Scene Parcing Network (PSPNet) была представлена китайским Университетом Гонконга и победила в конкурсе ImageNet Challenge 2016 года. Первой задачей PSPNet было улучшение модели полностью сверточной сети (FCN). Благодаря модулю Pyramid Pooling Module (PPM) нейронная сеть способна улавливать как глобальные, так и локальные особенности, что особенно важно для сложных изображений (рис. 5).
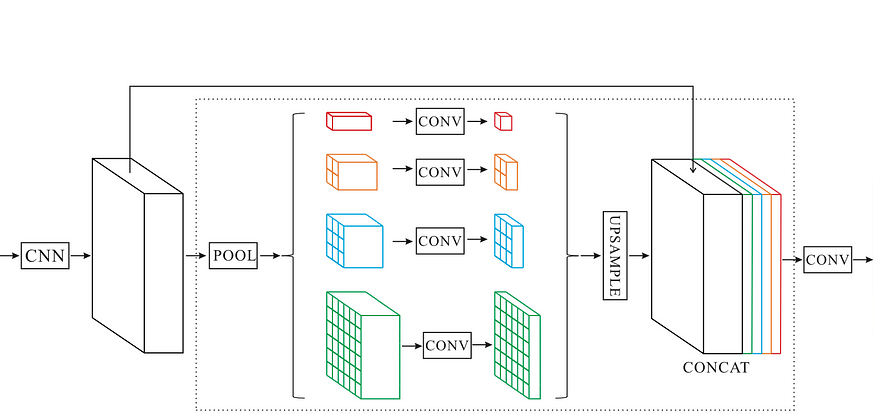
Сначала, данные проходят через так называемый backbone, то есть любая модель сверточной нейросети, которая извлечет все возможные признаки. В своем проекте я использовал ResNet-18, которая довольна легкая, но при этом эффективна. Для более верной интерпретации, в коде PSPNet в качестве основной функцией является только один остаточный блок, а не сразу два, как это было в LinkNet. По итогу, на выходе мы получаем 512 извлеченных карт признаков. Во всех остаточных блоках, кроме двух первых, шаг равен 2 и 1. Так же, здесь присутствует входной блок, как и в LinkNet. В результате вся сверточная нейросеть ResNet-18 выглядит так:
def pspnet_backbone(inputs):
x = Conv2D(64, (7, 7), strides=2, use_bias=False, padding='same', kernel_initializer='he_normal')(inputs)
x = BatchNormalization()(x)
x = ReLU()(x)
x = MaxPooling2D((3, 3), strides=2, padding='same')(x)
x = resnet18_residual_block(x, 64, stride=1)
x = resnet18_residual_block(x, 64, stride=1)
x = resnet18_residual_block(x, 128, stride=2)
x = resnet18_residual_block(x, 128, stride=1)
x = resnet18_residual_block(x, 256, stride=2)
x = resnet18_residual_block(x, 256, stride=1)
x = resnet18_residual_block(x, 512, stride=2)
x = resnet18_residual_block(x, 512, stride=1)
return xДалее идет самая уникальная деталь данной нейросети, а именно Pyramid Pooling Module. Этот модуль разбивает карты признаков на несколько слоев с разным разрешением, каждый из которых извлекает определенные особенности. PPM создает пирамиду, выполняя Average Pooling с различным разрешением (1x1, 2x2, 3x3, 6x6). На уровне 1x1 (red_pixel) карта признаков сжимается в одно значение для каждого канала, чтобы обеспечить глобальный контекст изображения. На уровне 6x6 (green_pixel), с другой стороны, извлекаются более локализованные признаки.
В x.shape[1:3] извлекаются пространственные размеры входного тензора (высота и ширина). Это необходимо для последующего увеличения (upsampling), чтобы привести выходы из каждого пула к тому же размеру, что и исходный тензор. GlobalAveragePooling2D() сводит все пространственные измерения каждого канала к одному значению (обычно вычисляя их среднее), поэтому на выходе получается 1×1 карта признаков для каждого канала, но оно возвращает тензор формы (batch, channels). Чтобы этот результат можно было обработать через Conv2D, требуется восстановить пространственные оси, поэтому используется Reshape(), превращающий его в форму (batch, 1, 1, channels). В случае же с AveragePooling2D() тензор не изменяется, так что дополнительные преобразования не требуются. Для достижения баланса между глобальными и локальными признаками при их последующем объединении на всех уровнях используются сверточные слои с ядром 1х1. Затем на каждом уровне происходит увеличение до размера исходной карты признаков, которые мы подавали на вход PPM. Ну и в самом конце функция concatenate() объединяет все карты признаков разных уровней, и в том числе, которые получены на выходе сверточной нейросети ResNet-18.
def pyramid_pooling_module(x):
input_shape = x.shape[1:3]
red_pixel = GlobalAveragePooling2D()(x)
red_pixel = Reshape((1, 1, -1))(red_pixel)
red_pixel = Conv2D(64, (1, 1), padding='same', use_bias=False)(red_pixel)
red_pixel = BatchNormalization()(red_pixel)
red_pixel = UpSampling2D(size=input_shape, interpolation='bilinear')(red_pixel)
yellow_pixel = AveragePooling2D(pool_size=(2, 2))(x)
yellow_pixel = Conv2D(64, (1, 1), padding='same', use_bias=False)(yellow_pixel)
yellow_pixel = BatchNormalization()(yellow_pixel)
yellow_pixel = UpSampling2D(size=2, interpolation='bilinear')(yellow_pixel)
blue_pixel = AveragePooling2D(pool_size=(3, 3))(x)
blue_pixel = Conv2D(64, (1, 1), padding='same', use_bias=False)(blue_pixel)
blue_pixel = BatchNormalization()(blue_pixel)
blue_pixel = UpSampling2D(size=3, interpolation='bilinear')(blue_pixel)
green_pixel = AveragePooling2D(pool_size=(6, 6))(x)
green_pixel = Conv2D(64, (1, 1), padding='same', use_bias=False)(green_pixel)
green_pixel = BatchNormalization()(green_pixel)
green_pixel = UpSampling2D(size=input_shape, interpolation='bilinear')(green_pixel)
return concatenate([x, red_pixel, yellow_pixel, blue_pixel, green_pixel])Остался финальный блок. В нем размер объединенных признаков увеличивается до исходного размера изображения. В функции Upsmapling2D() необходим IMG_HEIGHT // ppm.shape[1], IMG_WIDTH // ppm.shape[2], тем самым она будет динамически давать коэффициент, на который нужно увеличить карту по вертикали и горизонтали, чтобы восстановить исходное разрешение. Остальные действия точно такие же, как и в предыдущих нейросетях. Оригинальная статья: Pyramid Scene Parsing Network
final_feature = UpSampling2D(size=(IMG_HEIGHT // ppm.shape[1], IMG_WIDTH // ppm.shape[2]), interpolation='bilinear')(ppm)
outputs = Conv2D(n_classes, (3,3), padding='same', use_bias=False, kernel_initializer='he_normal')(final_feature)
outputs = BatchNormalization()(outputs)
outputs = Softmax()(outputs)Вот и подошел к концу наш экскурс по трем одним из самых популярных нейросетей для семантической сегментации. Мы узнали основные элементы подготовки и преобразования входных данных, а также рассмотрели важные блоки нейросетей U-Net, LinkNet, PSPNet. Если вы нашли неточности или что-то непонятное, то можете обязательно пишите. Это мне поможет в будущем писать публикации еще более качественно. Посмотреть полноценные версии всех файлов можно на моем GitHub.
Всем спасибо за внимание!
Автор: BJ10




