Этот пост является логическим продолжением моего первого поста о Байесовских методах, который можно найти тут.
Я бы хотел подробно рассказать о том, как проводить анализ на практике.
Как я уже упоминал, наиболее популярным средством, используемым для Байесовского анализа, является язык R с пакетами JAGS и/или BUGS. Для рядового пользователя различие в пакетах состоит в кросс-платформенности JAGS (на собственном опыте убедился в наличии конфликта между Ubuntu и BUGS), а также в том, что в JAGS можно создавать свои функции и распределения (впрочем, необходимость в этом у рядового пользователя возникает нечасто, если вообще возникает). Кстати, отличным и удобным IDE для R является, например, RStudio.
Но в этом посте я напишу об альтернативе R — Python с модулем pymc.
В качестве удобного IDE для Python могу предложить spyder.
Я отдаю предпочтение Python потому, что, во-первых, не вижу смысла изучать такой не слишком распространенный язык, как R, только для того, чтобы посчитать какую-то задачку, касающуюся статистики. Во-вторых, Python с его модулями, с моей точки зрения, ничем не уступает R в простоте, понятности и функциональности.
Я предлагаю решить простую задачу нахождения коэффициентов линейной зависимости данных. Подобная задача оптимизации параметров довольно часто встречается в самых разных областях знаний, поэтому я бы сказал, что она весьма показательна. В конце мы сможем сравнить результат Байесовского анализа и метода наименьших квадратов
Установка ПО
Прежде всего нам надо установить Python (для тех, кто этого еще не сделал). Я не пользовался Python 3.3, но с 2.7 все точно работает.
Затем нужно установить все необходимые модули для Python: numpy, Matplotlib.
При желании вы можете также установить дополнительные модули: scipy, pyTables, pydot, IPython и nose.
Все эти установки (кроме самого Python) проще делать через setuptools.
А теперь можно установить собственно pymc (его можно также поставить через setuptools).
Подробный гайд по pymc можно найти тут.
Формулировка задачи
У нас есть данные, полученные в ходе гипотетического эксперимента, которые линейно зависят от некоторой величины x. Данные поступают с шумом, дисперсия которого неизвестна. Необходимо найти коэффициенты линейной зависимости.
Решение
Сперва мы импортируем модули, которые мы установили, и которые нам понадобятся:
import numpy
import pymc
Затем нам надо получить наши гипотетические линейные данные.
Для этого мы определяем, какое количество точек мы хотим иметь (в данном случае 20), они равномерно распределены на отрезке [0, 10], и задаем реальные коэффициенты линейной зависимости. Далее мы накладываем на данные гауссов шум:
#Generate data with noise
number_points = 20
true_coefficients = [10.4, 5.5]
x = numpy.linspace(0, 10, number_points)
noise = numpy.random.normal(size = number_points)
data = true_coefficients[0]*x + true_coefficients[1] + noise
Итак, у нас есть данные, и теперь нам надо подумать, как мы хотим проводить анализ.
Во-первых, мы знаем (или предполагаем), что наш шум гауссов, значит, функция правдоподобия у нас будет гауссова. У нее есть два параметра: среднее и дисперсия. Так как среднее шума равно нулю, то среднее для функции правдоподобия будет задаваться значением модели (а модель у нас линейная, поэтому там два параметра). Тогда как дисперсия нам неизвестна, следовательно, она будет еще одним параметром.
В результате у нас есть три параметра и гауссова функция правдоподобия.
Мы ничего не знаем о значениях параметров, поэтому a priori предположим их равномерно распределенными с произвольными границами (эти границы можно отодвигать сколь угодно далеко).
При задании априорного распределения, мы должны указать метку, по которой мы будем узнавать об апостериорных значениях параметров (первый аргумент), а также указать границы распределения (второй и третий аргументы). Все вышеперечисленные аргументы являются обязательными (еще есть дополнительные аргументы, описание которых можно найти в документации).
sigma = pymc.Uniform('sigma', 0., 100.)
a = pymc.Uniform('a', 0., 20.)
b = pymc.Uniform('b', 0., 20.)
Теперь надо задать нашу модель. В pymc сушествует два наиболее часто используемых класса: детерминистический и стохастический. Если при заданных входных данных можно однозначно определить значение(-я), которое(-ые) модель возвращает, значит, это детерминистическая модель. В нашем случае при заданных коэффициентах линейной зависимости для любой точки мы можем однозначно определить результат, соответственно это детерминистическая модель:
@pymc.deterministic(plot=False)
def linear_fit(a=a, b=b, x=x):
return a*x + b
И наконец задаем функцию правдоподобия, в которой среднее — это значение модели, sigma — параметр с заданным априорным распределением, а data — это наши экспериментальные данные:
y = pymc.Normal('y', mu=linear_fit, tau=1.0/sigma**2, value=data, observed=True)
Итак, весь файл model.py выглядит следующим образом:
import numpy
import pymc
#Generate data with noise
number_points = 20
true_coefficients = [10.4, 5.5]
x = numpy.linspace(0, 10, number_points)
noise = numpy.random.normal(size = number_points)
data = true_coefficients[0]*x + true_coefficients[1] + noise
#PRIORs:
#as sigma is unknown then we define it as a parameter:
sigma = pymc.Uniform('sigma', 0., 100.)
#fitting the line y = a*x+b, hence the coefficient are parameters:
a = pymc.Uniform('a', 0., 20.)
b = pymc.Uniform('b', 0., 20.)
#define the model: if a, b and x are given the return value is determined, hence the model is deterministic:
@pymc.deterministic(plot=False)
def linear_fit(a=a, b=b, x=x):
return a*x + b
#LIKELIHOOD
#normal likelihood with observed data (with noise), model value and sigma
y = pymc.Normal('y', mu=linear_fit, tau=1.0/sigma**2, value=data, observed=True)
Теперь я предлагаю сделать небольшое теоретическое отступление и обсудить, что же все-таки делает pymc.
С точки зрения матеметики, нам нужно решить следующую задачу:
p(a, b, sigma | Data) = p(Data | a, b, sigma)*p(a, b, sigma) / p(Data)
Т.к. a, b и sigma независимы, то мы можем переписать уравнение в следующем виде:
p(a, b, sigma | Data) = p(Data | a, b, sigma)*p(a)*p( b)*p(sigma) / p(Data)
На бумаге задача выглядит очень просто, но, когда мы ее решаем численно (при этом мы должны думать еще и о том, что мы хотим численно решить любую задачу такого класса, а не только с определенными типами вероятностей), то тут возникают трудности.
p(Data) — это, как обсуждалось в моем предыдущем посте, константа.
p(Data | a, b, sigma) нам определенно задана (то есть при известных a, b и sigma мы можем однозначно рассчитать вероятности для наших имеющихся данных)
a вот вместо p(a), p( b) и p(sigma) у нас, по сути, имеются только генераторы псевдослучайных величин, распределенных по заданному нами закону.
Как из всего этого получить апостериорное распределение? Правильно, генерировать (делать выборку) a, b и sigma, а потом считать p(Data | a, b, sigma). В результате у нас получится цепочка значений, которая является выборкой из апостериорного распределения. Но тут возникает вопрос, каким образом мы можем сделать эту выборку правильно. Предположим, что наше апостериорное распределение имеет несколько мод («холмов») — тут возникает вопрос, как можно сгенерировать выборку, покрывающую все моды. То есть задача в том, как эффективно сделать выборку, которая бы «покрывала» все распределение за наименьшее количество итераций. Для этого есть несколько алгоритмов, наиболее используемый из которых MCMC (Markov chain Monte Carlo). Цепь Маркова — это такая последовательность случайных событий, в которой каждый элемент зависит от предыдущего, но не зависит от предпредыдущего. Я не стану описывать сам алгоритм (это может быть темой отдельного поста), но только отмечу, что pymc реализует этот алгоритм и в качестве результата дает цепь Маркова, являющуюся выборкой из апостериорного распределения. Вообще говоря, если мы не хотим, чтобы цепь была марковской, то нам просто надо ее «утоньшить», т.е. брать, например, каждый второй элемент.
Итак, мы создаем второй файл, назовем его run_model.py, в котором будем генерировать цепь Маркова. Файлы model.py и run_model.py должны быть в одной папке, иначе, в файл run_model.py нужно добавить код:
from sys import path
path.append("путь/к/папке/с/файлом/model.py/")
Вначале мы импортируем некоторые модули, которые нам пригодятся:
from numpy import polyfit
from matplotlib.pyplot import figure, plot, show, legend
import pymc
import model
polyfit реализует метод наименьших квадратов — с ним мы сравним результаты байесовского анализа.
figure, plot, show, legend нужны для того, чтобы построить итоговый график.
model — это, собственно, наша модель.
Затем мы создаем MCMC объект и запускаем выборку:
D = pymc.MCMC(model, db = 'pickle')
D.sample(iter = 10000, burn = 1000)
D.sample принимает два аргумента (на самом деле можно задать больше) — количество итераций и burn-in (назовем его «периодом разогрева»). Период разогрева — это количество первых итераций, которые обрезаются. Дело в том, что MCMC вначале зависит от стартовой точки (вот такое уж свойство), поэтому нам необходимо отрезать этот период зависимости.
На этом наш анализ закончен.
Теперь у нас есть объект D, в котором находится выборка, и который имеет различные методы (функции), позволяющие рассчитать параметры этой выборки (среднее, наиболее вероятное значение, дисперсию и пр.).
Для того, чтобы сравнить результаты, мы делаем анализ методом наименьших квадратов:
chisq_result = polyfit(model.x, model.data, 1)
Теперь печатаем все результаты:
print "nnResult of chi-square result: a= %f, b= %f" % (chisq_result[0], chisq_result[1])
print "nResult of Bayesian analysis: a= %f, b= %f" % (D.a.value, D.b.value)
print "nThe real coefficients are: a= %f, b= %fn" %(model.true_coefficients[0], model.true_coefficients[1])
Строим стандартные для pymc графики:
pymc.Matplot.plot(D)
И, наконец, строим наш итоговый график:
figure()
plot(model.x, model.data, marker='+', linestyle='')
plot(model.x, D.a.value * model.x + D.b.value, color='g', label='Bayes')
plot(model.x, chisq_result[0] * model.x + chisq_result[1], color='r', label='Chi-squared')
plot(model.x, model.true_coefficients[0] * model.x + model.true_coefficients[1], color='k', label='Data')
legend()
show()
Вот полное содержание файла run_model.py:
from numpy import polyfit
from matplotlib.pyplot import figure, plot, show, legend
import pymc
import model
#Define MCMC:
D = pymc.MCMC(model, db = 'pickle')
#Sample MCMC: 10000 iterations, burn-in period is 1000
D.sample(iter = 10000, burn = 1000)
#compute chi-squared fitting for comparison:
chisq_result = polyfit(model.x, model.data, 1)
#print the results:
print "nnResult of chi-square result: a= %f, b= %f" % (chisq_result[0], chisq_result[1])
print "nResult of Bayesian analysis: a= %f, b= %f" % (D.a.value, D.b.value)
print "nThe real coefficients are: a= %f, b= %fn" %(model.true_coefficients[0], model.true_coefficients[1])
#plot graphs from MCMC:
pymc.Matplot.plot(D)
#plot noised data, true line and two fitted lines (bayes and chi-squared):
figure()
plot(model.x, model.data, marker='+', linestyle='')
plot(model.x, D.a.value * model.x + D.b.value, color='g', label='Bayes')
plot(model.x, chisq_result[0] * model.x + chisq_result[1], color='r', label='Chi-squared')
plot(model.x, model.true_coefficients[0] * model.x + model.true_coefficients[1], color='k', label='Data')
legend()
show()
В терминале мы видим следующий ответ:
Result of chi-square result: a= 10.321533, b= 6.307100
Result of Bayesian analysis: a= 10.366272, b= 6.068982
The real coefficients are: a= 10.400000, b= 5.500000
Замечу, что, так как мы имеет дело со случайным процессом, те значения, которые вы увидите у себя, могут отличаться от вышеприведенных (кроме последней строки).
А в папке с файлом run_model.py мы увидим следующие графики.
Для параметра a:
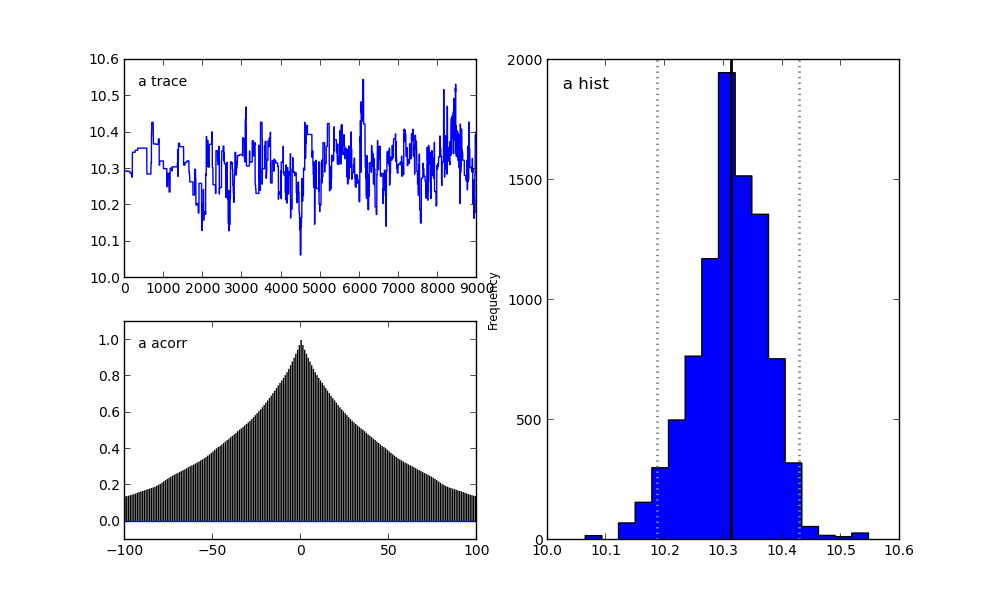
Для параметра b:
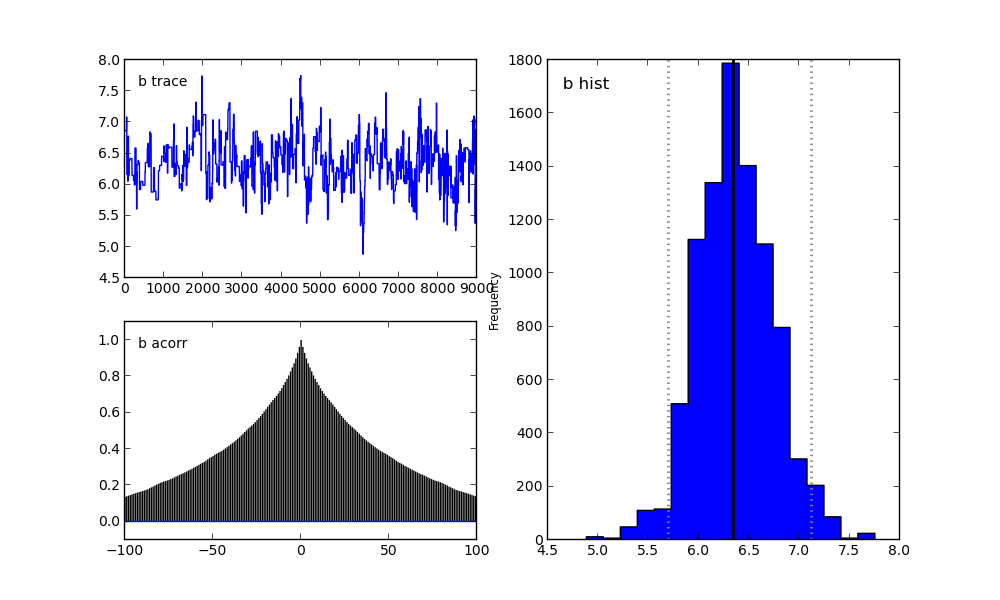
Для параметра sigma:
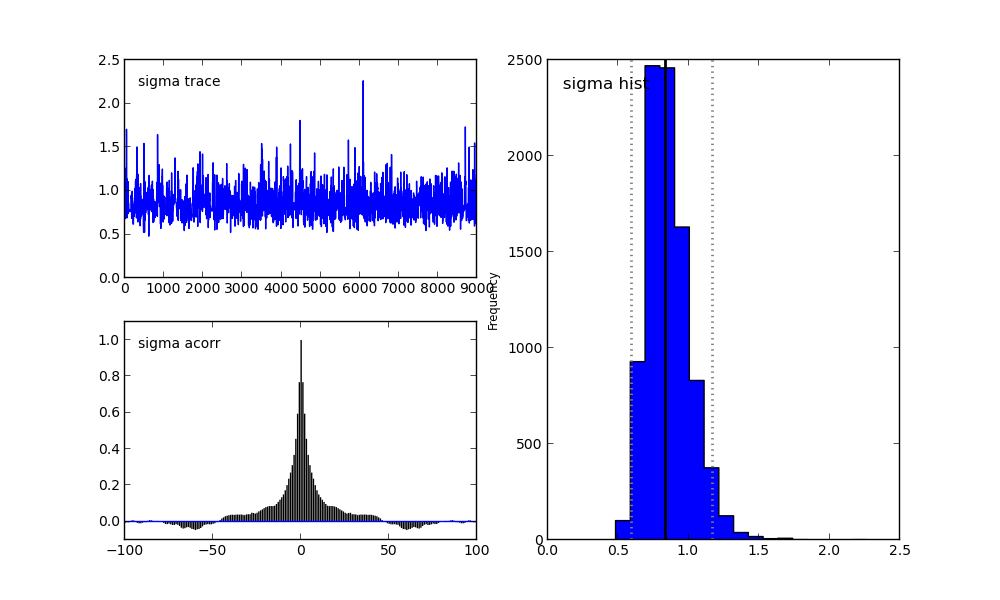
Справа мы видим гистограмму апостериорного распределения, а две картинки слева относятся к цепи Маркова.
На них я сейчас заострять внимание не буду. Скажу лишь, что нижний график — это график автокорреляции (подробнее можно прочитать тут). Он дает представление о сходимости MCMC.
А верхний график показывает след выборки. То есть он показывает, каким образом происходила выборка с течением времени. Среднее этого следа есть среднее выборки (сравните вертикальную ось на этом графике с горизонтальной осью на гистограмме справа).
В заключение я расскажу еще об одной интересной опции.
Если все же поставить пакет pydot и в файле run_model.py включить следующую строку:
pymc.graph.dag(D).write_png('dag.png')
То он создаст в папке с файлом run_model.py следующий рисунок:
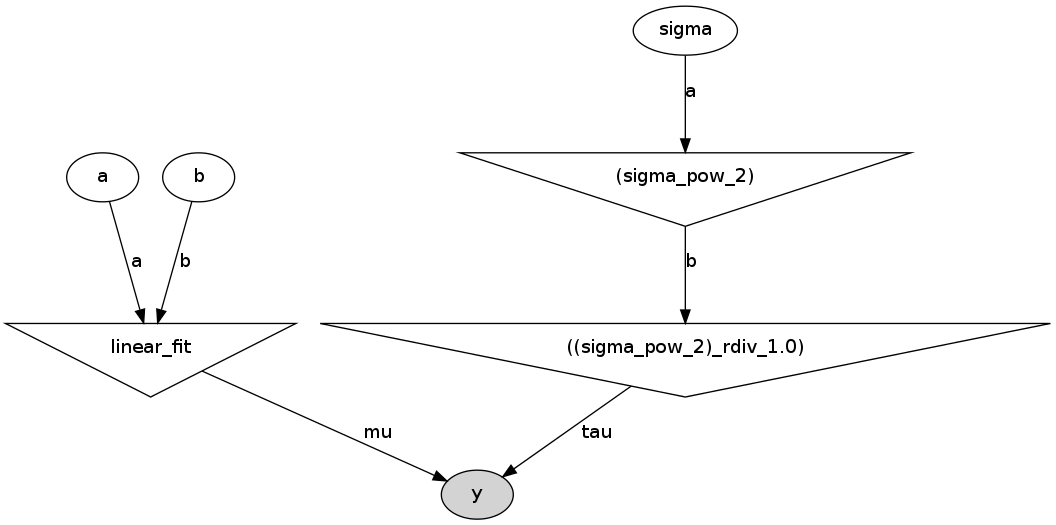
Это прямой ацикличный граф, представляющий нашу модель. Белые эллипсы показывают стохастические узлы (это a, b и sigma), треугольники — детерминистические узлы, а затемненный эллипс включает наши псевдоэкспериментальные данные.
То есть мы видим, что значания a и b поступают в нашу модель (linear_fit), которая сама по себе является детерминистским узлом, а потом поступают в функцию правдоподобия y. Sigma сначала задается стохастическим узлом, но так как параметром в функции правдоподобия является не sigma, а tau = 1/sigma^2, то стохастическое значение sigma сначала возводится в квадрат (верхний треугольник справа), и потом считается tau. И уже tau поступает в функцию правдоподобия, так же как и наши сгенерированные данные.
Я думаю, что этот граф весьма полезен как для объяснения модели, так и для самостоятельной проверки логики
модели.
Автор: Maxim_I






