Источник: https://mlr3book.mlr-org.com/
Привет!
В этом сообщении мы рассмотрим самый продуманный на сегодняшний день подход к машинному обучению на языке R — пакет mlr3 и экосистему вокруг него. Данный подход основан на «нормальном» ООП с использованием R6-классов и на представлении всех операций с данными и моделями в виде графа вычислений. Это позволяет создавать упорядоченные и гибкие пайплайны для задач машинного обучения, но на первых порах может показаться сложным и запутанным. Ниже постараемся внести определенную ясность и замотивировать к использованию mlr3 в ваших проектах.
Содержание:
- Немного истории и сравнение с конкурирующими решениями
- Технические детали: R6-классы и пакет data.table
- Основные составляющие ML-пайплайна в mlr3
- Настройка гиперпараметров
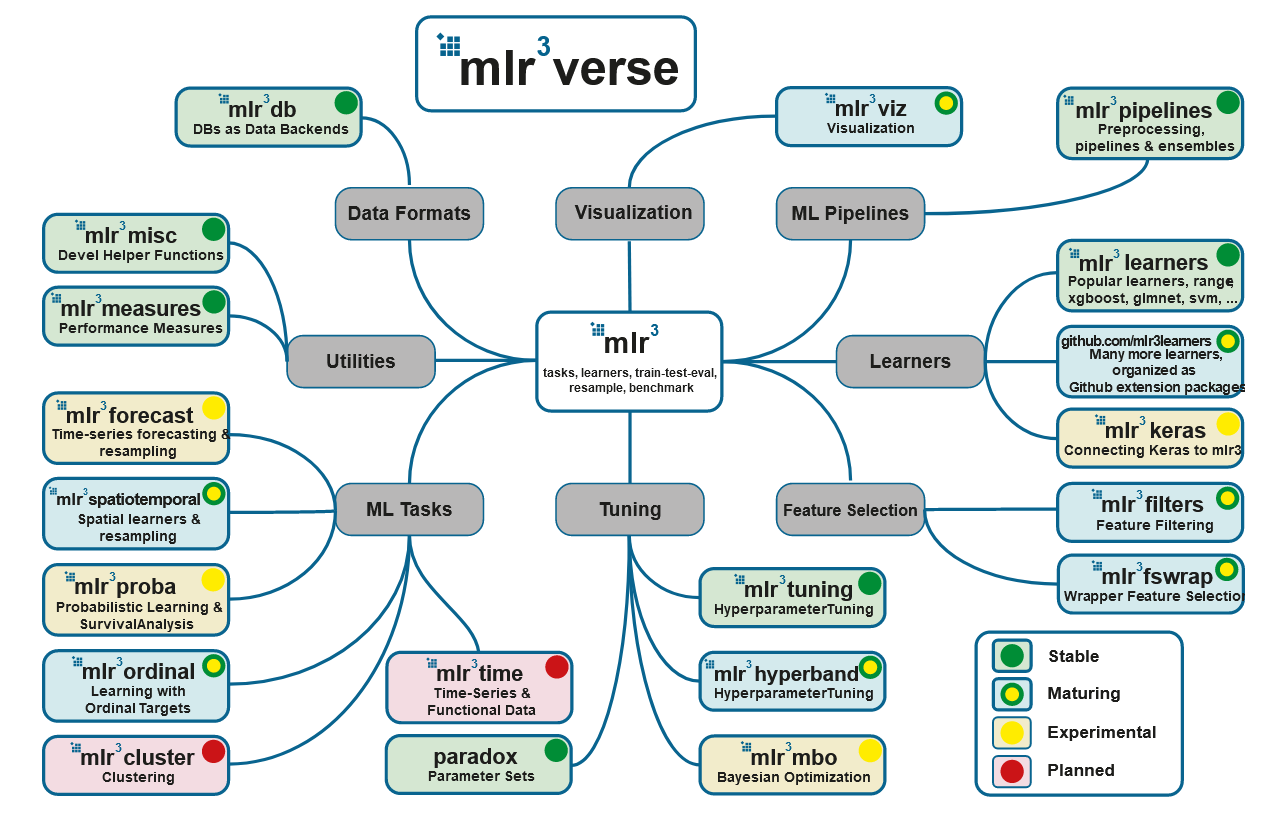
- Обзор экосистемы mlr3
- Пайпы и граф вычислений
1. Немного истории и сравнение с конкурирующими решениями
caret — старый, но не бесполезный
Пакет caret является первой реализацией инфраструктуры для построения моделей машинного обучения на R и одной из первых библиотек такого рода в целом (релиз на CRAN состоялся в 2007 году). В 2013 году по уже классическому на тот момент пакету была издана не менее классическая книга Applied Predictive Modeling, которую в комплекте с официальной документацией и сейчас можно рекомендовать в качестве вводного практического руководства по машинному обучению.
Преимущества:
- простота использования для стандартных задач (без экзотических схем кросс-валидации и многоуровневого стекинга);
- реализованы классические способы разбивки данных для (кросс-)валидации, функции предварительной обработки типа шкалирования, импутации и удаления коррелирующих признаков, метрики качества и методы отбора признаков;
- поддерживается огромное количество моделей, работать с которыми по отдельности без caret-овских оберток довольно неудобно из-за неунифицированных интерфейсов;
- достаточно разумный выбор настраиваемых гиперпараметров — например, для xgboost это оказывающие наибольшее влияние на качество параметры
nrounds,max_depth,eta,gamma,colsample_bytree,min_child_weightиsubsample.
Недостатки:
- первый минус является следствием последнего из перечисленных преимуществ — если хочется настраивать дополнительные гиперпараметры, придется написать свою обертку для соответствующей модели. Создание таких оберток является достаточно трудоемким;
- модели трактуются как алгоритмы машинного обучения без этапа предварительной обработки данных и создания признаков: этот этап выполняется на всех данных, а не внутри ресемплов при перекрестной проверке. Пакет recipes частично решает данную проблему, но об этом ниже;
- нет вложенной кросс-валидации (nested resampling), ограниченные возможности для создания ансамблей при помощи пакета caretEnsemble.
tidyverse strikes back
Своебразной работой над ошибками стало создание семейства пакетов под общей вывеской tidymodels, основными из которых являются recipes (отвечает за создание «рецептов» предварительной обработки данных, исполняемых внутри ресемплов с обучением на обучающей выборке и применением на обучающей и валидационной), rsample (обеспечивает различные варианты разбивки данных) и относительно новый tune (реализует собственно тюнинг гиперпараметров).
Преимущества:
- «рецепты» позволяют выполнять предварительную обработку данных внутри ресемплов, что является верным подходом для борьбы с переобучением;
- продвинутые методы предварительной обработки, в том числе реализованные в пакетах embed и textrecipes;
- можно настраивать любые гиперпараметры моделей, а не определенное разработчиками пакета их подмножество. Также можно настраивать гиперпараметры этапов предобработки (пакет tune);
- пакет workflows добавляет абстракцию для модели как комбинации «рецепта» и алгоритма машинного обучения.
Недостатки:
- чтобы работать с самими вариантами предобработки как с гиперпараметрами, возможностей пакета tune недостаточно. «Рецепт» нужно параметризировать, написав для этого функцию, а затем перебрать разные варианты предобработки при помощи цикла либо
apply/map-функции; - создание собственных этапов предобработки является исключительно запутанным и сложным для дебага. Например, для реализации кодирования средним или медианой пришлось написать 200 строк кода;
- вложенную кросс-валидацию и ансамбли нужно реализовывать вручную.
mlr3 vs все остальные
Пакет mlr3 и экосистема вокруг него также представляют собой попытку исправить недостатки как более раннего пакета mlr тех же авторов, так и рассмотренных выше caret и tidymodels. mlr подробно рассматривать не будем ввиду того, что его развитие было остановлено в пользу mlr3.
Преимущества:
- в основе лежат R6-классы, в качестве бекенда по умолчанию для табличных данных используется data.table;
- все процессы построения моделей объединены в граф вычислений. В составе этого графа можно задать любую схему перекрестной проверки и ансамблирования, перебрать разные модели с тюнингом гиперпараметров для каждой из них и разные варианты предобработки;
- вместо отдельных этапов с разными API для предобработки, создания признаков и обучения модели используется
learner— абстракция для модели как совокупности алгоритма машинного обучения и всех этапов трансформации данных; - модульность и относительная простота расширения.
Недостатки:
- стандартные проблемы пакетов на стадии активной разработки: не все фичи реализованы, местами не хватает примеров (этот недостаток активно исправляется), попадаются мертвые ссылки в документации;
- выбор поддерживаемых моделей пока что невелик.
2. Технические детали: R6-классы и пакет data.table
В основе экосистемы mlr3 лежат «нормальное» ООП, реализуемое путем использования R6-классов. R6-объекты являются изменяемыми, что позволяет работать с ними без копирования и перезаписи. Подробно изучить тему можно по официальной документации и книге Advanced R, мы же ограничимся кратким примером, позаиствованным из упомянутой книги.
Новый R6-класс создается вызовом функции R6Class():
library(R6)
Accumulator <- R6Class("Accumulator", list(
sum = 0,
add = function(x = 1) {
self$sum <- self$sum + x
invisible(self)
})
)Имя объекта должно совпадать с именем класса — в данном случае это "Accumulator".
У объектов есть метод new(), который позволяет создавать (или, как любят говорить настоящие программисты, инстанцировать) экземпляры класса:
x <- Accumulator$new() Функции, заданные внутри списка при определении класса, доступны как методы у экземпляров данного класса:
x$add(4)
x$sum
#> [1] 4R6-объекты передаются по ссылке:
y1 <- Accumulator$new()
y2 <- y1
y1$add(10)
c(y1 = y1$sum, y2 = y2$sum)
#> y1 y2
#> 10 10Поэтому для создания копий нужно вызывать метод clone() (указав clone(deep = TRUE) для рекурсивного копирования вложенных объектов):
y1 <- Accumulator$new()
y2 <- y1$clone()
y1$add(10)
c(y1 = y1$sum, y2 = y2$sum)
#> y1 y2
#> 10 0Это все, что нужно знать об R6 в контексте использования пакетов семейства mlr3.
Также целям устранения ненужного копирования и повышения скорости работы служит использование data.table в качестве бекенда по умолчанию (можно почитать перевод документации, недавний хабрапост Вокруг data.table и короткий обзор data.table: выжимаем максимум скорости при работе с данными в языке R). Киллер-фичей для использования в задачах машинного обучения является изменяемость таблиц data.table, позволяющая добавлять новые столбцы при помощи оператора := без перезаписи всей таблицы. Например, можно добавить столбец предсказанных значений к таблице с обучающей выборкой, не используя при этом 2х памяти относительно объема, занимаемого самой таблице. А при последовательном добавлении признаков в таблицу становится заметной еще и экономия по времени, и чем тяжелее таблица, тем экономия существеннее.
3. Основные составляющие ML-пайплайна в mlr3

Источник: https://mlr3book.mlr-org.com/
Минимальный пример решения задачи машинного обучения при помощи mlr3 выглядит следующим образом:
library(mlr3)
# Задача
task <- TaskClassif$new(id = "iris",
backend = iris,
target = "Species")
task
# <TaskClassif:iris> (150 x 5)
# * Target: Species
# * Properties: multiclass
# * Features (4):
# - dbl (4): Petal.Length, Petal.Width, Sepal.Length, Sepal.Width
# Модель
# learner_rpart <- mlr_learners$get("classif.rpart")
learner_rpart <- lrn("classif.rpart",
predict_type = "prob",
minsplit = 50)
learner_rpart
# <LearnerClassifRpart:classif.rpart>
# * Model: -
# * Parameters: xval=0, minsplit=50
# * Packages: rpart
# * Predict Type: prob
# * Feature types: logical, integer, numeric, factor, ordered
# * Properties: importance, missings, multiclass, selected_features, twoclass, weights
# Гиперпараметры модели
learner_rpart$param_set
# ParamSet:
# id class lower upper levels default value
# 1: minsplit ParamInt 1 Inf 20 50
# 2: minbucket ParamInt 1 Inf <NoDefault>
# 3: cp ParamDbl 0 1 0.01
# 4: maxcompete ParamInt 0 Inf 4
# 5: maxsurrogate ParamInt 0 Inf 5
# 6: maxdepth ParamInt 1 30 30
# 7: usesurrogate ParamInt 0 2 2
# 8: surrogatestyle ParamInt 0 1 0
# 9: xval ParamInt 0 Inf 10 0
# Обучение
learner_rpart$train(task, row_ids = 1:120)
learner_rpart$model
# n= 120
#
# node), split, n, loss, yval, (yprob)
# * denotes terminal node
#
# 1) root 120 70 setosa (0.41666667 0.41666667 0.16666667)
# 2) Petal.Length< 2.45 50 0 setosa (1.00000000 0.00000000 0.00000000) *
# 3) Petal.Length>=2.45 70 20 versicolor (0.00000000 0.71428571 0.28571429)
# 6) Petal.Length< 4.95 49 1 versicolor (0.00000000 0.97959184 0.02040816) *
# 7) Petal.Length>=4.95 21 2 virginica (0.00000000 0.09523810 0.90476190) *В процессе участвуют две сущности: задача (task) и модель (learner).
Задача создается как экземпляр соответствующего класса (TaskClassif для классификации, TaskRegr для регрессии и т.д.) путем вызова метода new(). Нужно указать идентификатор задачи id, таблицу с данными backend и целевую переменную target; в случае бинарной классификации положительный класс задается параметром positive. Стандартные задачи можно получить с использованием альтернативного синтаксиса: mlr_tasks$get("iris") или tsk("iris").
Модель извлекается из списка mlr_learners при помощи метода get() и затем обучается посредством вызова метода train(), в который передается наша задача task и строки выборки, участвующие в обучении. Но удобнее создавать модели с использованием синтаксического сахара: lrn("classif.rpart", predict_type = "prob", minsplit = 50). В этом случае можно сразу задать настройки модели (predict_type = "prob") и значения гиперпараметров (minsplit = 50). После создания модели их тоже легко поменять: learner_rpart$predict_type <- "prob", learner_rpart$param_set$values$minsplit = 50.
Обученную модель используем для предсказания на новых данных при помощи метода predict_newdata():
# Предсказание на новых данных
preds <- learner_rpart$predict_newdata(newdata = iris[121:150, ])
preds
# <PredictionClassif> for 30 observations:
# row_id truth response prob.setosa prob.versicolor prob.virginica
# 1 virginica virginica 0 0.0952381 0.90476190
# 2 virginica versicolor 0 0.9795918 0.02040816
# 3 virginica virginica 0 0.0952381 0.90476190
# ---
# 28 virginica virginica 0 0.0952381 0.90476190
# 29 virginica virginica 0 0.0952381 0.90476190
# 30 virginica virginica 0 0.0952381 0.90476190Добавим кросс-валидацию с разбивкой на 5 фолдов:
cv10 <- rsmp("cv", folds = 5)
resample_results <- resample(task, learner_rpart, cv10)
# INFO [09:37:05.993] Applying learner 'classif.rpart' on task 'iris' (iter 1/5)
# INFO [09:37:06.018] Applying learner 'classif.rpart' on task 'iris' (iter 2/5)
# INFO [09:37:06.042] Applying learner 'classif.rpart' on task 'iris' (iter 3/5)
# INFO [09:37:06.074] Applying learner 'classif.rpart' on task 'iris' (iter 4/5)
# INFO [09:37:06.098] Applying learner 'classif.rpart' on task 'iris' (iter 5/5)
resample_results
# <ResampleResult> of 5 iterations
# * Task: iris
# * Learner: classif.rpart
# * Warnings: 0 in 0 iterations
# * Errors: 0 in 0 iterations
# Список других вариантов (кросс-)валидации:
as.data.table(mlr_resamplings)
# key params iters
# 1: bootstrap repeats,ratio 30
# 2: custom 0
# 3: cv folds 10
# 4: holdout ratio 1
# 5: repeated_cv repeats,folds 100
# 6: subsampling repeats,ratio 30Оценим качество полученной модели. Для этого вызовем метод score() у объекта с ресемплами resample_resuts, передав ему список из двух метрик — accuracy "classif.acc" и classification error "classif.ce". Метрики также хранятся в списке, элементы которого извлекаются методом get(): mlr_measures$get("classif.ce"). Но мы вновь воспользуемся синтаксическим сахаром в виде функции msrs():
resample_resuts$score(msrs(c("classif.acc", "classif.ce")))[, 5:10]
# Выводим часть столбцов
# resampling resampling_id iteration prediction classif.acc classif.ce
# 1: <ResamplingCV> cv 1 <list> 0.8666667 0.13333333
# 2: <ResamplingCV> cv 2 <list> 0.9666667 0.03333333
# 3: <ResamplingCV> cv 3 <list> 0.9333333 0.06666667
# 4: <ResamplingCV> cv 4 <list> 0.9666667 0.03333333
# 5: <ResamplingCV> cv 5 <list> 0.9333333 0.066666674. Настройка гиперпараметров
Осталось самое главное — произвести настройку гиперпараметров модели. Тут все немного сложнее, и вызовом одного метода дело не ограничится.
Прежде всего зададим пространство для перебора значений гиперпараметров. Этим функционалом заведует отдельный пакет paradox:
library("paradox")
searchspace <- ParamSet$new(list(
ParamDbl$new("cp", lower = 0.001, upper = 0.1),
ParamInt$new("minsplit", lower = 1, upper = 10)
))
searchspace
# ParamSet:
# id class lower upper levels default value
# 1: cp ParamDbl 0.001 0.1 <NoDefault>
# 2: minsplit ParamInt 1.000 10.0 <NoDefault> Мы сконструировали новый объект класса ParamSet, определив в нем диапазон проверяемых значений для числового параметра cp и целочисленного параметра minsplit; остальные гиперпараметры нашей модели rpart оставим по умолчанию.
Важным моментом является то, что объект searchspace не содержит в себе никаких реальных значений. Эти значения будут сгенерированы при вызове метода tune() объекта класса Tuner. Границы диапазонов всегда включаются в набор значений. Количество проверяемых вариантов задается числом resolution, если нужно равное количество для всех гиперпараметров, или именованным вектором param_resolutions, если нужно разное количество для разных гиперпараметров. Кроме того, фактическое число проверяемых комбинаций ограничивается бюджетом на вычисления, но об этом чуть позже.
Функция generate_design_grid() позволяет получить таблицу значений гиперпараметров, по которой будет проводиться перебор:
generate_design_grid(searchspace,
param_resolutions = c("cp" = 2, "minsplit" = 3))
# <Design> with 6 rows:
# cp minsplit
# 1: 0.001 1
# 2: 0.001 5
# 3: 0.001 10
# 4: 0.100 1
# 5: 0.100 5
# 6: 0.100 10Также реализованы другие способы генерации сетки значений: generate_design_random() для случайной выборки из диапазона и generate_design_lhs() для создания дизайна эксперимента методом латинского гиперкуба.
Как уже было сказано, фактическое число проверяемых комбинаций можно ограничить. Для этого существуют различные варианты Terminator-ов, реализующие ограничения по времени, количеству проверямых моделей (мы используем именно его), достижению целевого качества или выходу на плато. Для дальнейшей работы понадобится пакет mlr3tuning:
library("mlr3tuning")
evals20 <- term("evals", n_evals = 20)
evals20
# <TerminatorEvals>
# * Parameters: n_evals=20
# Другие варианты
as.data.table(mlr_terminators)
# key
# 1: clock_time
# 2: combo
# 3: evals
# 4: model_time
# 5: none
# 6: perf_reached
# 7: stagnationОбъединим все ингредиенты в один объект класса TuningInstance:
tuning_instance <- TuningInstance$new(
task = TaskClassif$new(id = "iris",
backend = iris,
target = "Species"),
learner = lrn("classif.rpart",
predict_type = "prob"),
resampling = rsmp("cv", folds = 5),
measures = msr("classif.ce"),
param_set = ParamSet$new(
list(ParamDbl$new("cp", lower = 0.001, upper = 0.1),
ParamInt$new("minsplit", lower = 1, upper = 10)
)
),
terminator = term("evals", n_evals = 20)
)
tuning_instance
# <TuningInstance>
# * State: Not tuned
# * Task: <TaskClassif:iris>
# * Learner: <LearnerClassifRpart:classif.rpart>
# * Measures: classif.ce
# * Resampling: <ResamplingCV>
# * Terminator: <TerminatorEvals>
# * bm_args: list()
# * n_evals: 0
# ParamSet:
# id class lower upper levels default value
# 1: cp ParamDbl 0.001 0.1 <NoDefault>
# 2: minsplit ParamInt 1.000 10.0 <NoDefault> Создадим тюнер — объект класса Tuner, реализующий ту или иную стратегию перебора значений гиперпараметров:
tuner <- tnr("grid_search",
resolution = 5,
batch_size = 2)
# Другие варианты
# as.data.table(mlr_tuners)
# key
# 1: design_points
# 2: gensa
# 3: grid_search
# 4: random_searchМы указали resolution = 5, что для двух гиперпараметров означает проверку 25 комбинаций. Но фактически будет проверено лишь 20 случайным образом выбранных комбинаций, поскольку мы задали terminator = term("evals", n_evals = 20). batch_size — неудачно выбранное название параметра, определяющего количество параллельно обучаемых моделей. Параллелизация в mlr3 — отдельная большая тема, выходящая за пределы данной статьи.
Заслуживает внимания тюнер tnr("design_points"): он позволяет передать созданную заранее таблицу со значениями гиперпараметров, что зачастую удобнее генерации из диапазонов (особенно если нужно перебрать значений на логарифмической шкале — без готовой таблицы придется использовать достаточно громоздкий механизм преобразования параметров, который в mlr3 тоже есть).
Наконец, запустим процесс:
result <- tuner$tune(tuning_instance)
result
# NULLКак видим, result не содержит ничего. Это потому, что вызов tuner$tune() приводит к изменению объекта tuning_instance:
tuning_instance$result
# $tune_x
# $tune_x$cp
# [1] 0.001
#
# $tune_x$minsplit
# [1] 5
#
#
# $params
# $params$xval
# [1] 0
#
# $params$cp
# [1] 0.001
#
# $params$minsplit
# [1] 5
#
#
# $perf
# classif.ce
# 0.04
result <- tuning_instance$archive(unnest = "params")
result[order(classif.ce), c("cp", "minsplit", "classif.ce")]
# cp minsplit classif.ce
# 1: 0.00100 5 0.04000000
# 2: 0.00100 3 0.04000000
# 3: 0.00100 8 0.04000000
# 4: 0.00100 1 0.04000000
# 5: 0.00100 10 0.04666667
# 6: 0.02575 10 0.06000000
# 7: 0.07525 5 0.06000000
# 8: 0.02575 8 0.06000000
# 9: 0.02575 3 0.06000000
# 10: 0.05050 1 0.06000000
# 11: 0.07525 3 0.06000000
# 12: 0.07525 1 0.06000000
# 13: 0.05050 3 0.06000000
# 14: 0.02575 5 0.06000000
# 15: 0.05050 5 0.06000000
# 16: 0.05050 8 0.06000000
# 17: 0.10000 3 0.06000000
# 18: 0.10000 8 0.06000000
# 19: 0.05050 10 0.06000000
# 20: 0.10000 1 0.06000000
library(ggplot2)
ggplot(result,
aes(x = cp, y = classif.ce, color = as.factor(minsplit))) +
geom_line() +
geom_point(size = 3)

Рассмотрим подробнее, что именно происходит после вызова метода tune():
Tunerиспользует как минимум один набор значений гиперпараметров (он может использовать несколько наборов в параллельном режиме в зависимости от значения параметраbatch_size);- для каждого набора значений гиперпараметров модель
Lernerобучается на задачеTaskсогласно заданной схеме ресемплов. Результаты сохраняются в объекте классаResampleResult(совокупность таких объектов хранится в объектеBenchmarkResult); Terminatorпроверяет, не исчерпался ли бюджет на вычисления. Если нет, снова переходим к пункту 1, и так до тех пор, пока бюджет не закончится;- определяется набор значений гиперпараметров с наилучшим качеством модели;
- сохраняются значения гиперпараметров и полученные метрики качества, усредненные по ресемплам (другие варианты агрегирования метрики можно задать при ее создании, например,
msr("classif.ce", aggregator = "median").
Дополнительную информацию о результатах обучения моделей можно получить из объекта tuning_instance$bmr, имеющего класс BenchmarkResult, при помощи его метода score() или функции as.data.table(tuning_instance$bmr). Что происходит на уровне отдельных ресемплов, можно понять, используя аналогичный метод для объектов ResampleResult из таблицы tuning_instance$archive():
tuning_instance$archive()[1, resample_result][[1]]$score()[, 4:9]
# learner_id resampling resampling_id iteration prediction classif.ce
# 1: classif.rpart <ResamplingCV> cv 1 <list> 0.06666667
# 2: classif.rpart <ResamplingCV> cv 2 <list> 0.16666667
# 3: classif.rpart <ResamplingCV> cv 3 <list> 0.03333333
# 4: classif.rpart <ResamplingCV> cv 4 <list> 0.03333333
# 5: classif.rpart <ResamplingCV> cv 5 <list> 0.00000000Например, можем добавить к таблице значения метрики качества на каждом ресемпле:
res <- tuning_instance$archive(unnest = "params")
res[, ce_resemples := lapply(resample_result, function(x) x$score()[, classif.ce])]5. Обзор экосистемы mlr3
С основными пакетами мы уже знакомы: это mlr3, mlr3tuning и paradox. Вся экосистема представлена на заглавной картинке и в списке, а основные пакеты можно поставить при помощи мета-пакета mlr3verse:
# install.packages("mlr3verse")
library(mlr3verse)
## Loading required package: mlr3
## Loading required package: mlr3db
## Loading required package: mlr3filters
## Loading required package: mlr3learners
## Loading required package: mlr3pipelines
## Loading required package: mlr3tuning
## Loading required package: mlr3viz
## Loading required package: paradox- mlr3db позволяет подключать dbplyr в качестве бекенда вместо data.table.
- mlr3filters содержит алгоритмы отбора признаков, в том числе на основе встроенных в модели метрик важности признаков (пользоваться ими нужно с осторожностью).
- mlr3learners является коллекцией моделей для регрессии (
regr.glmnet,regr.kknn,regr.km,regr.lm,regr.ranger,regr.svm,regr.xgboost) и классификации (classif.glmnet,classif.kknn,classif.lda,classif.log_reg,classif.multinom,classif.naive_bayes,classif.qda,classif.ranger,classif.svm,classif.xgboost). Дополнительные модели можно найти в отдельных пакетах. - mlr3pipelines содержит пайпы (pipelines), из которых строится вычислительный граф. Кроме того, в версии на гитхабе есть и целые вычислительные графы, которых пока нет в пакете на CRAN, так что лучше поставить именно ее:
remotes::install_github("https://github.com/mlr-org/mlr3pipelines"). - mlr3tuning был рассмотрен выше.
- mlr3viz служит для визуализации, в том числе отвечает за отрисовку вычислительных графов.
- mlr3measures — пакет с ~40 метриками качества. В состав mlr3verse не входит, нужно ставить руками.
Следите за страницами по представленным ссылкам, список пакетов будет пополняться.
6. Пайпы и граф вычислений
Про пайпы (pipelines) можно было бы написать много, но много уже написали разработчики, поэтому постараемся максимально кратко изложить наиболее принципиальные для практического использования моменты.
Все операции — отбор признаков, преобразования, само обучение модели — абстрагируются в виде пайпов. Для моделей есть PipeOpLearner(), для отбора признаков — PipeOpFilter(), для всех остальных преобразований — PipeOp(). Мы используем синтаксический сахар (функция po()) для всех трех случаев:
pca <- po("pca")
filter <- po("filter",
filter = mlr3filters::flt("variance"),
filter.frac = 0.5)
learner_po <- po("learner",
learner = lrn("classif.rpart"))Пайпы последовательно соединяются в граф при помощи оператора %>>%:
graph <- pca %>>% filter %>>% learner_po
graph$plot()

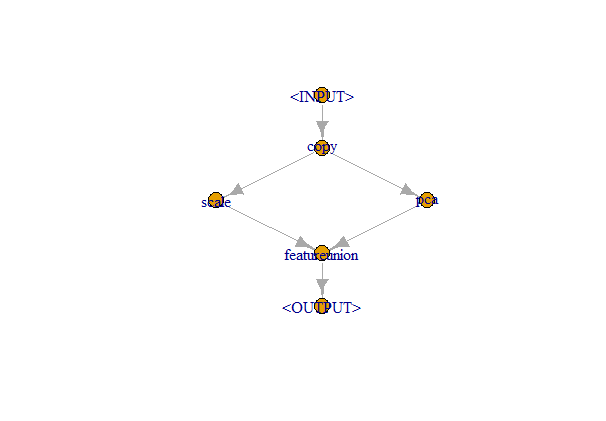
У пайпов есть входы и выходы. Для графов с более сложной структурой придется явно указывать, какой выход к какому входу последующего пайпа подключать:
gr <- Graph$new()$
add_pipeop(mlr_pipeops$get("copy", outnum = 2))$
add_pipeop(mlr_pipeops$get("scale"))$
add_pipeop(mlr_pipeops$get("pca"))$
add_pipeop(mlr_pipeops$get("featureunion", innum = 2))
gr$
add_edge("copy", "scale", src_channel = 1)$
add_edge("copy", "pca", src_channel = "output2")$
add_edge("scale", "featureunion", dst_channel = 1)$
add_edge("pca", "featureunion", dst_channel = 2)
gr$plot(html = FALSE)
Как сделать пайп из модели, мы уже видели (po("learner", learner = lrn("classif.rpart"))). В свою очередь, граф целиком можно сделать моделью:
glrn <- GraphLearner$new(graph)
glrn
# <GraphLearner:pca.variance.classif.rpart>
# * Model: -
# * Parameters: variance.filter.frac=0.5, variance.na.rm=TRUE, classif.rpart.xval=0
# * Packages: -
# * Predict Type: response
# * Feature types: logical, integer, numeric, character, factor, ordered, POSIXct
# * Properties: importance, missings, multiclass, oob_error, selected_features, twoclass,
# weightsПолучившийся объект относится к классам GraphLearner и Learner. Его можно использовать так же, как и рассмотренные выше простые Learner-ы, например:
resample(tsk("iris"), glrn, rsmp("cv"))
# INFO [17:17:00.358] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 1/10)
# INFO [17:17:00.615] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 2/10)
# INFO [17:17:00.881] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 3/10)
# INFO [17:17:01.087] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 4/10)
# INFO [17:17:01.303] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 5/10)
# INFO [17:17:01.518] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 6/10)
# INFO [17:17:01.716] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 7/10)
# INFO [17:17:01.927] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 8/10)
# INFO [17:17:02.129] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 9/10)
# INFO [17:17:02.337] Applying learner 'pca.variance.classif.rpart' on task 'iris' (iter 10/10)
# <ResampleResult> of 10 iterations
# * Task: iris
# * Learner: pca.variance.classif.rpart
# * Warnings: 0 in 0 iterations
# * Errors: 0 in 0 iterationsТретьего дня была реализована невиданная ранее фича, которая обсуждалась в issue How to deal with different preprocessing steps as hyperparameters:
gr <- pipeline_branch(list(pca = po("pca"), nothing = po("nop")))
gr$plot()
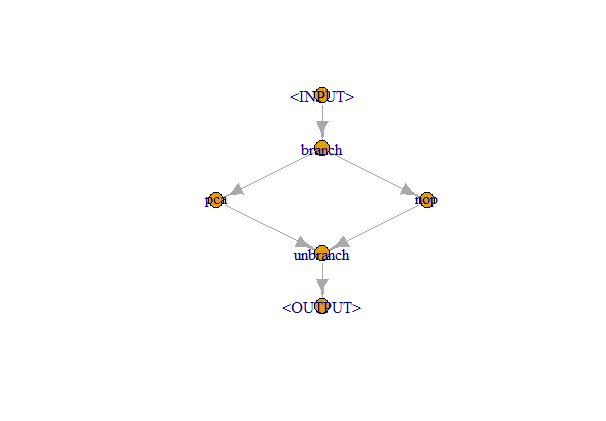
Рассмотренные в первом разделе caret и tidymodels так не умеют!
Надеюсь, данный пост был полезен и зародил интерес к дальнейшему изучению и использованию фреймворка mlr3. Подробнее можно почитать в книге mlr3 book и в галерее примеров.
Автор: Андрей Огурцов






