В этой статье мы рассмотрим как свернуть одну спираль в РНК. Для понимания нужно прочитать все предыдущие части От белков к РНК, Мат. критерии, Как уменьшить число поворотов цепи?, Как оценить ход сворачивания односпиральной РНК?, Ограничение оптимизирующих методов в играх с противником и без. Если ранее у нас все шло как по маслу, то здесь мы столкнемся с серьезной проблемой. Может кто-то подскажет как её решить.
Повторим рисунок вторичной структуры рибозима который мы будем сворачивать.
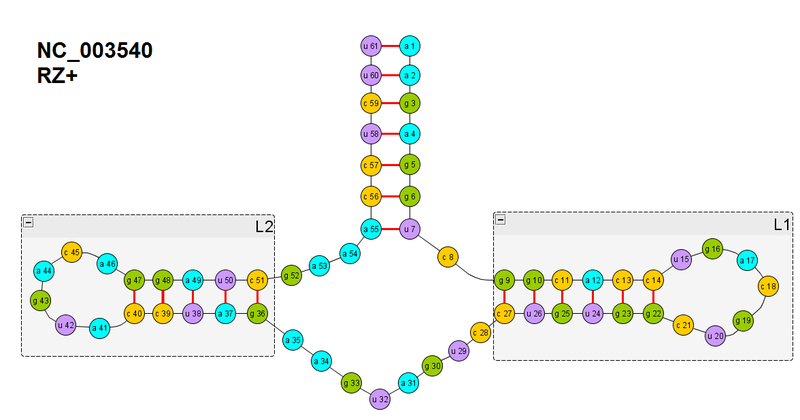
Здесь уделим внимание спирали L1. В предыдущей части мы ввели целевую функцию для образования водородных связей между одной парой нуклеотидов (назовем её F(x-y), где x-y пара нуклеотидов). Здесь же нам надо образовать целых шесть: между нуклеотидами 14-22, 13-23, 12-24, 11-25, 10-26, 9-27. Когда мы их образуем цепь РНК свернется в спираль сама собой -> таково геометрическое расположение атомов, которые образуют водородные связи.
Казалось бы мы можем взять и использовать такую целевую функцию:
Fo = F(14-22) + F(13-23) + F(12-24) + F(11-25) + F(10-26) + F(9-27)
Но получится так, что связи на конце спирали, т.е. между нуклеотидами 10-26, 9-27 будут образовываться быстрее. Почему? Ну в общем-то понятно, длина цепи для концов больше, возможностей состыковаться больше, вероятность быстрее найти нужные повороты больше.
Но чем это мешает? Дело в том, как только образуется водородная связь, атомы подходят так близко, что им становится затем так сложно найти поворот, чтобы наоборот отдалится друг от друга — велика вероятность столкновения. И это не физика — это геометрия в тех ограничения о которых я писал ранее.
Так вот, если почти образовались связи 10-26, 9-27, то при условии «не развалить эти связи», а наша целевая функция это предполагает, это почти сводит к нулю возможность образовать затем связи в начале цепи, т.е. между нуклеотидами 14-22, 13-23.
Конечно это связано с расчетами, а не с природой. В природе все хорошо.
Но как вращается цепь. В игре FoldIt — есть такая возможность — инструмент Rebuild. Он закрепляет конец цепи, и начинает вращать нуклеотиды. При этом если скажем проворачиваем 18 нуклеотид, то все нуклеотиды до него 1-17 остаются неподвижными. Но после поворота 18 нуклеотида обязательно изменят свое положение все остальные дальше, т.е. 19-61. Причем чем они дальше от 18-го тем сильнее они поменяют расположение.
Так вот, если мы образовали связь 9-27, и желаем образовать связь 14-22, то на это влияют повороты внутри петли 15, 16, 17, 18, 19, 20, 21… допустим поворот Х в 18 нуклеотиде сильно уменьшает целевую функцию F(14-22), но он одновременно разрывает связь 9-27, и целевая функция F(9-27) очень сильно возрастает. Понятно, что при общей целевой функции Fo мы такой поворот никогда не осуществим, а значит не сможем двигаться к намеченной цели.
В игре FoldIt есть такая возможность заморозить нуклеотиды. И тогда инструмент Rebuild вращает цепь только не замороженных нуклеотидов. Например, он может заморозить нуклеотиды 9-27, т.е. там где мы образовали связь, и вращать внутри между точками 9 — 27, не сдвигая их. Как он это делает? Прошу подскажите.
Я же вышел из этой проблемы изменением целевой функции Fo. Он видна на слайде

Идея очень простая — регулировать коэффициентом сжатия какие пары водородных связей нужно образовать в начале, а какие позже. При коэффициенте =1, эта функция превращается просто в сумму как и в Fo.
Это существенно улучшает сворачивание спирали. Но все равно могут быть зацикливания, особенно на малых расстояниях. Тогда приходится менять коэффициент сжатия, так или иначе разрывать связи и повторять процесс. Это уже делается в ручную, и автоматизировать невозможно.
Автор: tac