Это продолжение статьи Часть №1. Введение в биовычисления по сворачиванию. От белков к РНК. Здесь мы опишем ковалентные и водородные связи математически. Посмотрим какие углы мы будем вращать у РНК для сворачивания. И прикоснемся к вопросу «а в чем трудность то?»
Вначале два слайда:
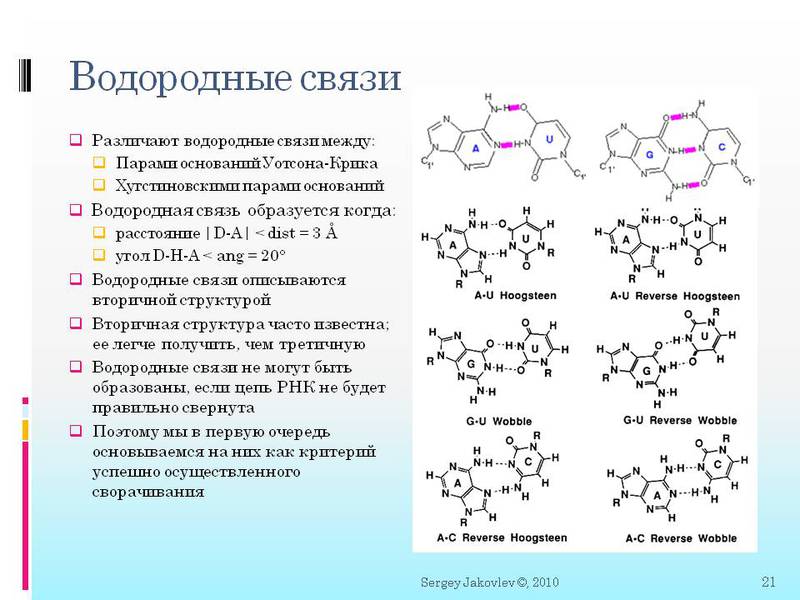

тут вы видите более строгое определение ковалентных и водородных связей. И то как они описываются математически. Далее нам будет важно:
1. Рассчитать появились ли запрещенные ковалентные связи дольше, чем сделать собственно поворот и рассчитать десяток водородных связей. Поэтому это делается только тогда, когда по всем остальным критериям нас устраивает найденное состояние. Считайте, что есть просто функция GetFullCovalentBond(), которая говорит нам есть ли хоть одна запрещенная ковалентная связь. Если есть, идем считать дальше и такие состояния не учитываем. Хотя сравнительно в игре FoldIt столкновения достаточно быстро просчитываются и избавляются от этих состояний из «плохих» состояний. В этом есть некоторый смысл. Но увы, у меня нет алгоритма как это нормально делать, в общем пока без этого.
2. Важно, что между молекулами образуется сразу или две или три (в зависимости от типа) водородных связи. Далее мы будем говорить в контексте «между нуклеотидами образовалась водородная связь» — означает, что образовались все две/три связи.
3. Вначале меня очень напрягало, что могут быть нестандартные водородные связи. Т.е. не как в ДНК только G с C и A с T (а в РНК с U). Могут быть разные сюрпризы. Например, G c U, а так-же тройные связи A c двумя U одновременно.
4. Еще раз скажу, так как это важно — водородная связь образуется тогда когда определенные атомы находятся на расстоянии <3 ангстрем друг от друга и под определенным углом <20 градусов. Все координаты атомов рассчитываются в ангстремах. И существует функция ScoreGroup(int N1, int N2), куда подается два номера нуклеотидов (молекул) в цепи, и получаем ответ на сколько они близки для создания всех нужных водородных связей. Тут важно то, что ответ не просто да/нет, а как функция приближения к «да». Что именно за функция будем говорить в последующих статьях.
Теперь как мы можем поворачивать цепь. Начнем с поворотов только одного нуклеотида:

Цепь поворачивается только путем поворотов 9 торсионных углов. Ни как по другому она двигаться не может — запрещено биологией (энергией). 6 углов находятся в так называемой главной цепи, то что на рисунках в прошлой статье показывается сплошной линией. Тут обозначены греческими буквами начиная с альфа. Еще три угла в боковой цепи, сильно между собой зависимы, т.е. нельзя их поворачивать независимо — поэтому далее мы будем называть это углом с номером 7 — но на самом деле там будет комбинация из трех углов. Боковая цепь — это радикал, т.е. то, чем собственно отличаются нуклеотиды a, u, c, g — а главная цепь (12 атомов из 28-33) у них одинакова.
Если мы имеем только один нуклеотид — понятно, что по 6 углам можно вращаться почти без запретов. Да и по 7 -му очень мало запретов. Ничего не мешает. Если же уже есть цепь из 2 нуклеотидов, то появляются запреты — атомы одного нуклеотида могут наскочит на атомы другого. Но так как связка не большая (возьмите бусы — два рядом стоящих шарика не сильно то могут с собой столкнуться при вращении — длина веревочки мала между ними) — то запретов тоже не много. Но если взять 6 и более уже запретов может быть много.
И тут уже поворот по одному углу на 0.1 градуса может позволить повернутся по другому углу, например для образования водородной связи, и без этого не получится.
В чем трудность? Просчитать все комбинации скажем за сутки можно только для цепи из 2-3 нуклеотидов, для 4 уже нужна неделя-месяц, и далее годы :)
Но не так уж все углы коррелированны между собой. Есть те которые мало друг на друга влияют. Но никто не знает какие это в данном случае.
Но за шаг взять и установить нужные углы не получается, поэтому хочется разработать процедуру итерационную, чтобы шаг за шагом получать все лучшие и лучшие состояние. Но я делал такой эксперимент. Брал всего 3 нуклеотида (биологически вырожденное состояние). И делал повороты вначале первого нуклеотида, находил лучшие состояние, затем второго фиксируем лучшие, затем третьего (одна итерация — просчет скажем 1500+1500+1500 поворотов) фиксируем лучшие… и дальше и дальше. Алгоритм со временем находит такое состояние с оценкой -9.41.
Если же сделать так — поворачиваем первый нуклеотид на 1500 поворотов, затем для каждого из 1500 вариантов поворачиваем второй на 1500, затем для комбинации первого-второго (1500*1500 вариантов) поворачиваем третий на 1500 вариантов поворота. Мы получим действительно полный перебор из примерно 3 млрд. вариантов, и получим лучшие состояние -13.87.
Но путь полнейшего перебора закрыт, а хотелось бы свести к варианту локального приближения (первый вариант). Как это сделать мы поговорим в следующей части.
Для углубленного понимания дам две ссылки:
1. О деталях описанного выше эксперимента
2. О свободе колебаний нуклеотидов в спирали РНК, там хорошо видно, что законы максимально нелинейные и никакой простой логики не существует.
Автор: tac






