Этот проект был задуман давно. Лет 5 назад я считал, что многие результаты в геномике могут быть получены людьми далекими от биологии, коим я в полной мере являюсь. Конечно за это время я немного нахватался терминологии и немного узнал как работают специалисты. Но чем больше я узнавал как работают специалисты тем большие отторжение это у меня вызывало. Я считаю, что они явно много незаслуженно усложняют в результате чего не простая область становится не проходимой. В то время как все достаточно просто и качественно можно сделать. И да я с ними пытаюсь конкурировать (конечно, только в определенной узкой области), как бы наивно это не выглядело.
Вся проблема этого проекта — это то, что я его единственный полноценный участник. Конечно, я успел со многими за это время поговорить и многие оказали реальное влияние на проект. Всем им спасибо. Понятно, что не коммерческий проект не сильно может рассчитывать на успех. Да, действительно за каждым научным проектом стоит солидные около миллионные вливания и команда серьезных ученных. У нас этого нет, а есть лишь гуманизм и энтузиазм.
Поэтому в первую очередь я нуждаюсь в советах от тех у кого есть опыт в стартапе подобных проектов на не коммерческой основе. Во вторую очередь, нужна собственно команда программистов (от знания биологии, при необходимости, я вас освобожу :) ). А сейчас я хотел бы найти таких энтузиастов, которые могли бы обеспечить работу (скажем скромно) домашней веб-страницы проекта (прошу писать мне на почту tac@inbox.lv или личными сообщениями хабра). И конечно, важен любой другой отклик и предложения.
А ниже я расскажу идею и то на что претендует проект, а также о текущих результатах, а они в худшем случае сравнимы с теми которые дают специалисты. Но я вполне самокритичен, поэтому всегда готов выслушать критику — желательно не в мой адрес, а в адрес проекта.
От идеи к компьютерным экспериментам
Сырую идею я излагать не буду, уже многое пройдено и было мной описано в прошлых статьях на хабре. Я опишу новый, что называется полноприводный эксперимент. Но вначале мне надо ввести вас в проблематику и затем понять как оценивать полученные результаты эксперимента.
Филогенетический сигнал
Здесь попробуем обсудить этот термин, на который обратил мое внимание один биолог.
При эволюционном происхождении животных от общего предка, считается что можно выстроить единую древовидную иерархическую структуру происхождения видов. При этом нет принципиальной разницы какие признаки брать за основу. Просто чем больше генов включается в анализ, тем меньше остается в дереве слабо обоснованных участков. В то же время если классифицируемые объекты не происходят от общего предка, то отсутствует единая древовидная иерархическая структура. Классификация таких объектов либо получается принципиально различной при использовании разных наборов признаков (генов), либо имеет принципиально не «древесный» вид.
А вот совпадение получаемых «деревьев» построенных по разным признакам якобы говорит нам о наличии «филогенетического сигнала». И чем меньше различия между деревьями, построенными по разным наборам генов, тем более сильный «филогенетический сигнал» мы имеем. Но что важно, обратное не верно.
Часто говорят, что этот сигнал действительно имеется и совпадает. Но это не совсем так, так мне попалась одна статья, которая несколько более критична на этот счет.
Во-первых они указывают, что:
Предполагается, что анализом множества генов можно усилить филогенетический сигнал до его превышения над шумом и добиться правильного разрешения конфликтов между различными генами. Но
[идут ряд частных примеров]
Все это говорит о том, что нынешние методы реконструкции филогении по большому числу генов не избавляют от артефактов, известных для единичных генов. Здесь точно так же могут сказываться допущения моделей эволюции, разница в скорости эволюции видов, ошибки выравнивания и выбора ортологов, недостаточная репрезентативность таксономической выборки. Для устранения артефактов мультигенного филогенетического анализа предлагается селекция данных, что, конечно, делает его не столь формальным. Таким образом, практика современной филогеномики показывает, что статистическая поддержка реконструкций филогении повышается с увеличением числа сравниваемых генов, однако высокий уровень статистической поддержки дерева в целом или его отдельных узлов не может служить показателем правильности филогенетической реконструкции.
И во вторых задаются вопросом:
Как же найти для проверки ген или нуклеотид, достойный безграничного доверия? Чем меньший геологический срок существовала стволовая группа, тем меньше вероятность, что выбранный наудачу ген будет нести синапоморфию, притом не подверженную гомоплазиям и реверсиям. Чтобы добыть наверняка выигрышный билет в лотерее, есть способ – скупить весь тираж. Учитывая скорость развития технологии секвенирования и компьютерной обработки, применительно к геномам это может через несколько лет показаться не такой глупой идеей. С другой стороны, если родственное сходство у видов большое, то оно обнаружится во многих генах из числа выбранных наудачу и даже, вероятно, в одном достаточно протяженном гене, как 18S или 28S рРНК.
Это что называется классика биологии. А теперь попробуем подумать над этим.
В предыдущих статьях на роль таких генов «достойных доверия» я предлагал и показывал, что получится если это будет ген тРНК. Этот ген ничем не хуже, чем рРНК, который сейчас пользуется «безграничным доверием». Но в этой статье я покажу далее, что будет если «скупить весь тираж». Но до этого надо разобраться, чем плох вариант когда «безграничным доверием» пользуется рРНК.
И оказывается, что дело совсем не в выборе того или иного гена или нуклеотидной последовательности. И правильно, что мечтают (но странно почему не делают) о сравнении по большому множеству генов. Дело именно в методе. А он имеет статистическую природу, и те кто немного более трезво на это смотрят признают как выше в статье наличие проблем «Здесь точно так же могут сказываться допущения моделей эволюции, разница в скорости эволюции видов, ошибки выравнивания и выбора ортологов, недостаточная репрезентативность таксономической выборки».
Все это по отдельности ухудшает так или иначе филогенетический сигнал. Больше всего претензий к ошибкам выравнивания (не буду объяснять, что это почитайте Википедию по ссылке). Именно из-за этого приходится иметь дело со статистикой, и связанными с этим ошибками. Правильно сделать выравнивание, особенно для небольших последовательностей сейчас не умеют — оно реально не учитывает консервативность некоторых фрагментов. Для этого нужно учитывать водородные связи в третичной структуре — но этого обычно при выравнивании не делается.
Но рРНК, во-первых, длинная, во-вторых, по отдельности есть множество ошибок, но статистически они все же дают некий сигнал. А вот какого он качества мы ниже и разберемся на примере сравнения деревьев построенных по 16S рРНК и 23S рРНК (это самые длинные последовательности РНК из которых состоит рибосома). Такие деревья были получены в проекте The All-Species Living Tree. Но, а в третьих, сейчас пишут достаточное число статей по построению филогенетических деревьев, но вот такой вопрос как «анализ преобладания филогенетического сигнал над шумом» почему то не обсуждается.
А что в альтернативе?
Единственным вариантом, чтобы возразить на критику подобную выше приведенной («высокий уровень статистической поддержки дерева в целом или его отдельных узлов не может служить показателем правильности филогенетической реконструкции») состоит в том, чтобы перейти от статистических умозаключений, в которые здравый смысл не дает верить со 100% уверенностью, это перейти к умозаключениям детерминированного характера. А для этого надо избавится от выравнивания в анализе и выбрать те нуклеотидные последовательности, которые можно анализировать без выравнивания.
Я удивлен, но специалисты этой альтернативы не предлагают и не видят. Хотя она как минимум показывает более стабильные результаты. Почему? Вот с этим давайте и разберемся.
Ведь какое бы дерево я бы не дал в заключение доверие к нему будет не больше / не меньше, чем к другим деревьям. Но там строили специалисты (как например, в проекте The All-Species Living Tree), а тут вы скажите построил «шарлатан». И всегда найдутся возражения.
Точно также любой метод уязвим для критики, пока нет доверия к результатам. Поэтому нам нужен критерий правильности результатов. На такой критерий претендует стабильность «филогенетического сигнала».
Но прежде, чем его за такой выбрать — я хотел бы, чтобы читатель понимал бы почему вообще этот сигнал может быть не стабилен. Могут быть 3 причины:
1. Эволюция не идет по Дарвину, т.е. попросту у организмов нет общего предка и никогда его не было. Учитывая, во-первых, что сейчас есть явление горизонтального переноса, а во-вторых, что гипотеза о РНК-мире уже практически доказана, и тогда отдельные организмы могли возникать независимо друг от друга — эволюция по Дарвину на самом деле под большим вопросом. Поэтому тут мы просто согласимся с тем, что человеческому уму просто удобнее иерархически рассматривать происхождение видов и эволюция по Дарвину для нас просто удобный способ представления информации, аналогично рисованию графиков взамен текстовой информации.
2. Ошибки метода. Так например, выравнивание, к которому выше я высказал большое недоверие. Именно из-за неверного выравнивания происходит отклонение сигнала в большой мере.
3. Разное количество примеров в выборке.
Когда у нас есть влияние всех трех причин, мы не можем с полной уверенностью отличить полученный шум — это объективная причина или субъективная. Т.е. мы не можем сказать или проблема в нашем методе, проблема в нашей репрезентативности выборки или все же эволюция идет не совсем по Дарвину.
Исследователи очень легко могут сказать «а знаете наш метод работает идеально, выборка замечательная, а те мелкие погрешности которые вы видите — это просто так оно в природе и есть». Но во-первых, давайте будем измерять количественно погрешности. Во-вторых, заменим статистический подход на детерминированный. В третьих, сделаем анализ всего доступного для детерминированного подхода.
Преимущество детерминированного подхода
Чтобы продемонстрировать преимущество детерминированного подхода я предложу мысленный эксперимент. Его можно в реальности сделать экспериментально, но просто публика устанет от сухости изложения, и главное со времен Аристотеля мы знаем, что эксперимент не доказывает ничего в абсолютных категориях, а лишь позволяет сказать «на этих данных мы видим это, но это не значит, что не может быть по другому». А нам нужно судить именно в абсолютных категориях.
Итак мысленный эксперимент. Сравним статистический и детерминированный подход. В статистическом мы анализируем 1000 организмов на одном гене 16S рРНК, который имеет большую длину около 1600 символов (а это делается в подавляющем числе случаев при исследовании). Допустим у нас есть достоверный набор рРНК для всех 1000 организмов. Но для построения филогенетического дерева нам нужно сделать выравнивание. Но перед выравниванием разделим рРНК на две равные части и сделаем выравнивание и последующие построение дерева по первой и по второй части отдельно.
Так как выборка одинакова 3-я причина не оказывает влияния. На 1-ю причину мы договорились не ссылаться. Но очевидно, что выравнивание хоть в малой степени повлияет на вид дерева, т.к. там вычисляется некое эволюционное расстояние, а оно для разных частей хоть немного но будет отличаться. И в результате первое и второе дерево будет отличаться и это будет 2-я причина — ошибка метода.
Что имеем для детерминированного подхода. Тут мы ориентируемся на такие гены, которые в разных организмах полностью идентичны, но они не могут быть длинными, т.к. все длинное с большей вероятностью подвержено мутациям. Но вместо одного гена в 1600 символов, мы располагаем набором из 10-20 генов по 70-150 символов. Таким характеристикам, например, соответствует гены тРНК. Опять же предположим, что мы обладаем достоверным набором этих генов. Тогда вопрос заключается в следующим: если последовательности тРНК разделить на две части и построить два разных дерева — они совпадут или нет? Ответ: они совпадут на 100%. Это связано с тем, что при построении дерева на самом деле последовательности заменяются на идентификаторы, и далее все манипуляции происходят на основании лишь комбинаций генов. Поэтому если гены были правильно идентифицированы на основании половины последовательности, то дальше искажений не будет.
То есть в идеальных условиях и одинаковости выборки детерминированный подход имеет явное преимущество, и не имеет ошибок 2-го рода.
А дальше можно говорить об ошибках 3-го рода и как они влияют на филогенетический сигнал. Но мы должны понимать, что в детерминированном подходе мы только и имеем ошибки 3-го рода, а в статистическом, который принят сейчас повсеместно, мы не можем разделить влияние ошибок — «шума» 2-го и 3-го родов.
Собственно эксперимент
№1. Сравнение деревьев 16S и 23S
Итак нам надо сравнить между собой два дерева построенному по гену 23S рРНК и построенному по гену 23S рРНК являющиеся последним результатом проекта The All-Species Living Tree.
Но сравнивать можно лишь сравнимые вещи. И тут самое время поговорить о том как измерять величину ошибки 3-го рода, т.е. то как влияет величина выборки и её состав на результат. Специалисты тут бы нам предложили бы заняться статистическими изысканиями какие бы то распределениями вероятности, оценками смещения, дисперсии и т.п. мутными индексами и ничего не говорящими коэффициентами. В противовес мы должны сравнивать так, чтобы каждая цифра позволяла бы понять, что это означает.
Во-первых, формат филогенетических деревьев скрывает одну важную вещь — в них не отображается явно родитель, хотя он там есть как пересечение линий на одном уровне. По сути тут нам надо решить вопрос конвертации формата .newick например в формат .gml, т.е. получить полноценное дерево, где будут все предки иметь название.
Во-вторых, дело в том, что данных по гену 16S почти в 10 раз больше. И нам нужно убрать такие листьях деревьев, которые есть в дереве 16S, но их нет в дереве 23S, и наоборот. Только тогда мы получим то, что можно между собой сравнивать. Но после такого удаления (обрезания) «листьев» на дереве, которые мы не имеем возможности сравнить, могут остаться предполагаемые их предки и если они больше не имеют других «листьев», то их тоже надо убрать, чтобы они не засоряли дерево.
В-третьих, и это наиболее важно, выше сделанное описанное обрезание не решает всех проблем приведения дерева к одному знаменателю. Может возникнуть ситуация, что предок имеет только один лист, а этот предок имеет в свою очередь опять только одного предка, и так несколько раз. Т.е. в результате на дереве мы имеем «длинные нити». Все эти «единичные» предки не позволяют нам сравнить с другим деревом (23S) в которых этих предков нет, т.к. оно строилось на другой меньшей выборки, и естественно, что большая выборка заставляет предполагать большое число предков, чтобы более точно отобразить дивергенцию видов. Но чтобы это было сравнимо надо исключить таких «единичных» предков, а листья от них поднять на такой уровень, где есть предок больше чем с одним листом (т.е. где есть реальная дивергенция).
Этот процесс «подъема листьев в места дивергенции» снова оставит предков, которых можно исключить и этапы 2 и 3 нужно повторять пока не будут исключены все лишние предки.
Маленькая зарисовочка для понимания:
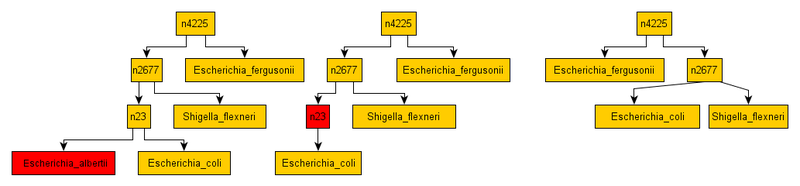
Справа вариант до всех манипуляций. По центру вариант, где обрезан лист «Escherichia_albertii», которого нет в сравниваемом дереве. Слева вариант, где убран излишний предок «n23». В реальности все более серьезно из 18000 узлов остаются нужных только 3000. Может также создаться впечатление, что убраны важные предки, но если их не убрать результат сравнения будет только хуже, так как в меньшем дереве «убранные» предки появится не могут, а сравнивать все же надо сравнимые вещи, а не «чайник с кастрюлей».
Теперь если строго подходить к сравнению, то совпадение деревьев это тогда, когда листья имеющие одного родителя в одном дереве имеют также одного родителя в сравниваемом дереве. И мы можем подсчитать число таких случаев. Но чтобы оценить близость надо также иметь некоторое распределение ошибок. Величину ошибки можно посчитать так. Если пара «листьев» в одном дереве имеет одного родителя, то в сравниваемом дереве мы находит их наименьшего общего предка LCA и считаем число промежуточных предков от одного листа до LCA и от второго до LCA — полученные числа складываем и наносим как точку на распределение ошибок.
В итоге имеем такой график, порядка 50% правильных случаев, а остальные несколько ошибочны, ошибка правда затухает.
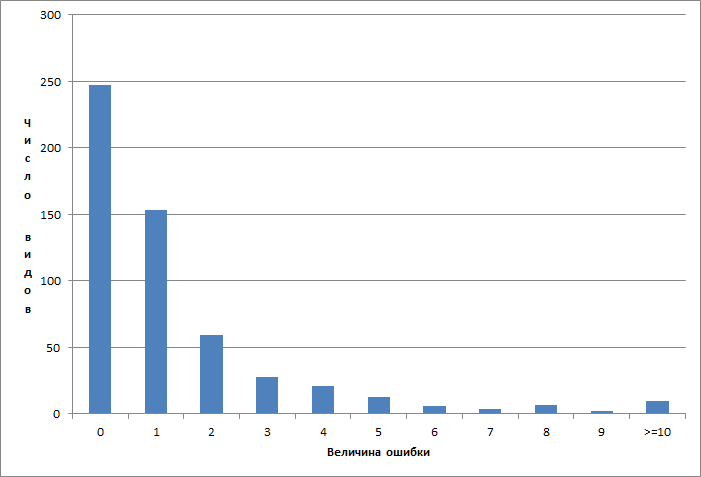
Как видим у специалистов все далеко от идеала, сигнал получается где-то на 50% зашумленный и далее хоть и пробивается какая-то закономерность, но не устойчивая. Поэтому есть что улучшать.
Продолжение следует…
Получается как-то длинно, поэтому результаты детерминированного подхода, я вынесу в отдельную статью. Там мы посмотрим на сколько удастся улучшить качество эволюционного дерева (филогенетического сигнала). Эксперимент в полной мере не закончен, но я надеюсь на лучшие :)
Автор: tac






