В предыдущей статье я не сильно утруждал себя детальным описанием идеи. Мне казалось она интуитивна понятна и элементарна. Но после дискуссии с Davidov понял, что идею не так то просто схватить. Дело в том, что сейчас классические представления в филогенетике строятся на одной догме, которая искажает мировоззрение биологов.
Когда мы строим эволюционное дерево — мы конечно же хотим знать в какой последовательности во времени видообразовывались виды. Но классическая филогенетика объявила, что это не научно ставить такой вопрос. И по сути расписалась в своем невежестве. Действительно, судить о времени видообразования, в то время как эволюционный процесс идет каждую минуту сложно. Но можно. Пояснить как это можно и призвано данное детализированное объяснение.
Идеальная точка расхождения видов
Рассматривать на пальцах, умозрительно будет не понятно, поэтому мы рассмотрим проблему на одном частном примере.
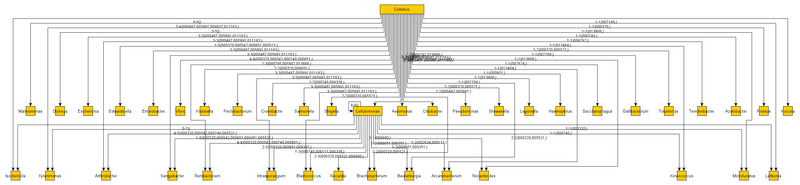
Обязательно смотрим пример в высоком разрешении.
Начнем с того, что такое вершина Cellvibrio. Cellvibrio — это один род протеобактерий. В роду есть несколько видов. Но секвенируют не вид, а одного из представителей вида — называется штамп (все равно, что взять геном конкретного человека — так получаем штамп вида). Теперь в данном примере получилось так, что сейчас секвенирован только один штамп Cellvibrio — вида Cellvibrio japonicus, а штамп назвали Ueda107. Поэтому на графе на вершине Cellvibrio нет связи с сами собой — нет информации.
Далее посмотрим на другую вершину Cellulomonas. Это другой род, и секвенированно два штампа разных видов. И около вершины есть связь с самим собой. И там стоят цифры 8-8. Это означает, что при сравнении этих штампов (в данном случае только двух) этого рода оказалось, что у них на 100% совпадают 8 разных тРНК.
Вообще в организме как правило около 20 тРНК (на каждую аминокислоту). Далее как правило организм содержит 2-3 вариации одной и той же тРНК. Но более древние организмы содержат даже не все 20 тРНК, и часто в одном варианте. Более молодые имеют большие разнообразие. Поэтому внутри рода если совпадают 40-60 тРНК — то можно с уверенностью говорить, что это сравнительно молодой род, и штампы к нему отнесены правильно. В данном случае у нас 8 — это маловато, но это как раз исключение, т.к. это один из древнейших организмов.
Откуда это следует (кроме как из малого числа совпадающих тРНК)?
В прошлой статье я давал полный граф систематики прокариот

Тут есть идеальная точка между самым большим черным пятном (там представители Гамма протеобактерий) и областью справа, где видны отдельные узлы. Это точка и есть вершина Cellvibrio.
Чем она примечательна? Сам род Cellvibrio относят к гаммапротеобактериям, но мы видим, что этот род имеет связи не только с кластером гаммапротеобактерий, но и с другим, например с вершиной Cellulomonas — а это род относят к типу Actinobacteria.
Встает вопрос: что более вероятно, что представители рода Cellvibrio агрегировали в себя тРНК из десятка гаммапротеобактерий и десятка других видов разных типов, например из Actinobacteria?
(Отдельно подчеркну, что даже совпадение одной тРНК это не случайное событие, а тут совпадает 5-7. )
Или же мы должны сделать вывод, что вид Cellvibrio japonicus существовал до того, как образовались протеобактерии и ряд типов, таких как Actinobacteria?
Естественно, что вероятность наличия одного вида вначале больше, чем агрегация сразу нескольких десятков видов в один вид.
Да, сейчас Cellvibrio japonicus — может отличаться, от Cellvibrio japonicus который был миллион лет назад, но за этот миллион лет его 5-7 тРНК не изменились вообще, в то время как белки и скажем рРНК подвергались мутациям.
Вот это один самый простой и яркий пример как можно судить о возрасте видов по отношению к другим.
Упорядочивание по возрасту
Но таких идеальных точек как мы видим на общем графе очень мало, это скорее случайность, когда так легко можно судить о том какой вид древнее, а какой более молодой.
Чаще идет наслоение одних родов на другие, и разобраться становится сложнее. Проследим это по первому графу. Вершина Cellvibrio имеет связи со всеми прочими вершинами, за исключением только одной Nocardia, которая связана так Cellvibrio — Cellulomonas — Nocardia. Т.е. мы можем здесь говорить, что род Nocardia более молодой по сравнению со всеми остальными, т.к. требует образования промежуточного рода Cellulomonas.
Как же проанализировать остальные связи?
Есть несколько способов в зависимости от желания детализации. Если посмотреть на подписи связей там мы увидим минимальное-максимальное число идентичных тРНК. Например, между родами Cellvibrio и Isoptericola (находится крайний слева) есть 6 идентичных тРНК. А с другой стороны между Cellvibrio и Kocuria (крайний справа) только 1 идентичная тРНК.
Таким образом можно говорить, что рода удаляются от Cellvibrio постепенно слева на право.
Это справедливо для первого ряда на 100%. И снова это позволяет говорить о времени образования родов.
Но со вторым рядом сложнее. Дело в том, что во втором ряде вершины имеют два входа от Cellvibrio и Cellulomonas, и становится не ясно от кого же из эти двух образовался следующий род.
Можно поступить статистически. Например, сравним две связи Cellvibrio — Isoptericola и Cellulomonas — Isoptericola. Первая имеет 6-6 идентичных тРНК, вторая 5-7. Значит можно сделать вывод, что от Cellvibrio к Cellulomonas передались 6 идентичных тРНК, и только потом образовался род Isoptericola которому передалась еще 1 тРНК, а в каком то виде потерялась 1 тРНК (итого передалось 5-7). И тогда связь Cellvibrio — Isoptericola опосредованна и ее можно удалить. И становится более точно видно кто от кого происходит во времени.
Есть и другие варианты. Там уже можно смотреть не статистически, а детально, есть ли конкретные тРНК, как предпосылка для последующего образования рода. Объясню подробнее на этом же примере в комментариях — если будет интерес.
Т.е. основная идея в том, что Целое не может образоваться, если еще в природе нет частей, из которых должно состоять целое.
Автор: tac
