Эволюционная систематика пытается определить родство и их близость различных организмов. Если не так давно об этом судили по внешним признакам организмов (морфологии если точнее), то теперь однозначно перешли к суждению путем сравнения геномов этих организмов.
Но ДНК в организме занимает большие объемы и сравнить по ней схожесть организмов сильно затруднительно. Кроме того ДНК постоянно эволюционирует. Поэтому биологи начали основываться на рибосомной рибонуклеиновой кислоте (рРНК), т.к. эти молекулы обнаружены у всех клеточных форм жизни, их функции связаны с важнейшим для организма процессом трансляции, первичная структура в целом характеризуется высокой консервативностью.
Считается, что особенностью рРНК является нахождение вне сферы действия отбора, поэтому данные молекулы эволюционируют в результате спонтанных мутаций, происходящих с постоянной скоростью, и накопление таких мутаций зависит только от времени. Таким образом, мерой эволюционного расстояния между организмами служит количество нуклеотидных замен в молекулах сравниваемых рРНК.
Известно, что в рибосомах прокариот и эукариот присутствуют 3 типа рРНК. Информационная емкость крупных молекул больше, но их труднее анализировать. Поэтому наиболее удобным оказался анализ молекул рРНК средней величины: 16S (~1600 нуклеотидов). Систематика основывается на расчете коэффициентов сходства сравниваемых организмов. Именно на основании анализа рРНК современная систематика выделяет три домена бактерии, археи и эукариоты, а так же на этом основывается систематика, бактерий и архей X издания Берги.
Вот такое положение дел в этой сфере на данный момент. Мной же была сделана попытка создать основы для несколько другой, если хотите альтернативной, систематики. Почему? Консервативность рРНК тем не менее не достаточно велика, консервативны лишь некоторые её части. А так как есть достаточно вариабельные части у рРНК, то приходится делать допущения и предполагать, где были разрывы и вставки отдельных фрагментов при мутации. А т.н. выравнивание сейчас делается с очень большой погрешностью.
В итоге, я пришел к выводу, что необходимо при сравнении геномных последовательностей сравнивать такие участки, которые вообще не подвергались мутациям, и которые абсолютно идентичны в разных организмах.
Смотрим, что из этого получилось.
Существуют ли участки ДНК, которые вообще не подвергаются мутациям на протяжении длительного времени?
Но есть ли такие участки, которые вообще не подвергались мутациям, и абсолютно идентичны в разных организмах? Оказывается есть. Целый ряд белков (их ДНК код) абсолютно одинаковы для многих видов, отнесенных к одному роду, или даже семейству. Но еще большей консервативностью обладают транспортные РНК (тРНК). В хромосоме бактерии, как правило, существуют все 20 видов тРНК, каждая из которых ответственна за транспортировку определенной аминокислоты к месту синтеза белка. И вот на основании их можно проследить эволюционную связь не только отдельных семейств, но и целых классов и даже типов.
В данном исследовании я основывался только на одной тРНК, которая транспортирует аланин (Ala tRNA). Поэтому выводы о эволюционной связи не претендуют на окончательный результат. Для этого нужно сопоставить результаты в отношении других тРНК. Но тем не менее, проделанное исследование позволяет наметить ряд положений о близости некоторых родов бактерий. Затем будет показано, что о близких эволюционных связях нельзя говорить в терминах филогенетического дерева. Это вызвано тем фактом, что существует горизонтальный перенос и конъюгация у бактерий, и тут требуются другие подходы.
Анализ геномов
В центре нашего внимания будет род Yersinia (Чумная палочка), но в результате исследования были рассмотрены и другие такие как Shewanella, Pseudomonas, Vibrio, Erythrobacter, Pseudoalteromonas, Photobacterium и ряд других (всего 109 локусов)
тРНК у бактерий, как правило, имеет константную длину 76 нуклеотидов, при этом антикодон располагается в позициях 34,35,36. Аланин в ДНК кодируется четырьмя кодонами: GCT, GCC, GCA, GCG. Поэтому потенциально возможны 4 вида Ala tRNA, с антикодонами AGC, GGC, TGC, CGC.
Но у подавляющего большинства рассматриваемых бактерий в геноме есть только по 2 вида Ala tRNA_GCA и Ala tRNA_GCС. Есть, конечно, и исключения.
Для анализа были использованы секвенированные геномы ДНК, которые имеются в базе NCBI. Все идентичные немутировавшие тРНК были помечены уникальным идентификатором (Id). С помощь написанной для анализа компьютерной программы, в полуавтоматическом режиме с ручной верификацией, был составлен список разнообразных видов Ala tRNA, и их расположение в том или ином секвенированном локусе.
Результаты
У всех рассматриваемых штаммов рода Yersinia (9 шт.), имеется в геноме одинаковые Ala tRNA_GCA с Id=00046 и Ala tRNA_GCС с Id=00043. На основании этого факта, можно сделать вывод, что действительно эти штаммы имеют сильную эволюционную связь, и поэтому их всех относят к роду Yersinia.
Сейчас род Yersinia относят к семейству Enterobacteriaceae. Но на основании проделанного анализа, в рамках сходства по Ala tRNA, эта связь малообоснованна.
Если посмотреть на классических представителей семейства Enterobacteriaceae, таких как Escherichia, Salmonella, Shigella, Citrobacter, Cronobacter, Klebsiella, Pectobacterium, то у всех них совершенно другие Ala tRNA. А именно у них Ala tRNA_GCA с Id=00011 и Ala tRNA_GCС с Id=00012. На этом основании мы и можем считать перечисленные роды семейства Enterobacteriaceae классическими представителями.
И только с родом Photorhabdus, по одной линии Ala tRNA_GCA с Id=00046, Yersinia имеет связь. Поэтому род Yersinia имеет черты от разных семейств. Такой род мы назовем переходным родом между различными семействами.
Итак, если с семейством Enterobacteriaceae род Yersinia связывает только одна конфигурация не мутировавшей Ala tRNA_GCA с Id=00046 (и то частично), то встает вопрос с каким семейством род Yersinia связан по другой конфигурации не мутировавшей Ala tRNA_GCС с Id=00043?
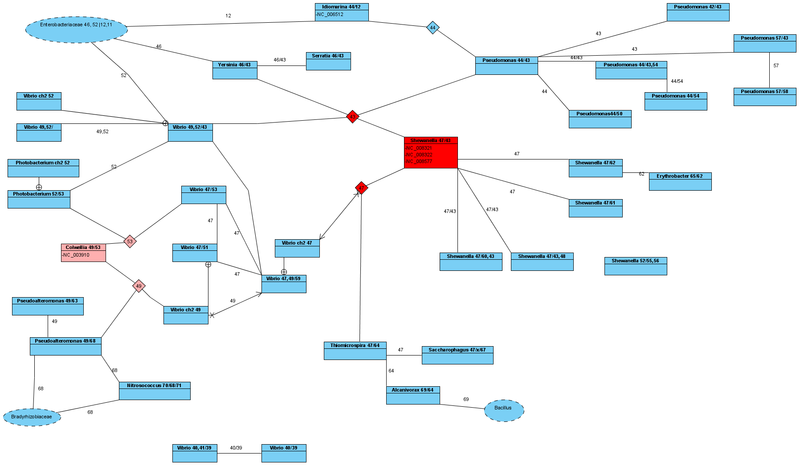
Оказывается наиболее прямой связью по этой линии является род Shewanella (семейство: Shewanellaceae, порядок: Alteromonadales, класс: гамма-протеобактерии). При этом объединяющая их линия Ala tRNA_GCС с Id=00043, является ключевой в эволюционном плане, так как присутствует так же еще у родов Pseudomonas и Vibrio. Все эти связи более близкие, чем следует из современной классификации, где эти роды объединены лишь на уровне класса.
В свою очередь некоторые представители рода Shewanella имеют одинаковые Ala tRNA_GCA с Id=00047 и Ala tRNA_GCС с Id=00043. Уже выяснив, что Yersinia и Shewanella имеют связь по одной линии (Id=00043), интересно с кем связан род Shewanella по другой линии (Id=00047). Оказывается, что эта линия также является достаточно ключевой в эволюционном плане. Она разветвляет происхождение далее и Ala tRNA_GCA с Id=00047 обладают так же представители родов Vibrio, Thiomicrospira, Saccharophagus.
Мы можем также проследить эти связи и далее (см. рисунок). Но уже сейчас понятно, чтобы систематизировать эти эволюционные связи, нужно несколько изменить подход к их описанию.
Выводы
Следует еще раз подчеркнуть, что все выводы сделанные в работе, базируются на анализе только tRNA Ala, и конечно для признания результатов нужна проверка по другим видам тРНК. Но тем ни менее уже сейчас мы можем сделать некоторые выводы и описать каким образом нужно изменить подходы к систематике таксонов.
Достаточную сложность вызывает определение какой из видов более эволюционно древний, а какой более молодой. Но если исходить из гипотезы, что биологический мир развивался от простого к сложному, то у нас есть по крайней мере один неоспоримый факт. Если бактерия имеет две хромосомы, то кажется очевидным, что она эволюционно более молода, чем имеющая одну.
Поэтому в рамках нашего исследования можно с уверенностью сказать, что род Vibrio более молодой, чем Shewanella или Yersinia. И тогда, раз Vibrio и Shewanella объединены идентичными Ala tRNAGCA с Id=00047, то с большой вероятностью, род Vibrio произошел от Shewanella и так произошла одна хромосома. После чего другие представители рода Vibrio произошли от рода Colwellia и так произошла вторая хромосома. В результате объединения этих хромосом в одном организме мы и можем говорить о роде Vibrio, который по одной линии произошел от Shewanella, а по другой от Colwellia.
Таким образом, мы должны говорить не о происхождении от одного предка, а о как минимум двух, а то и больше.
С однохромосомными бактериями сложнее определить направление эволюции (кто более молодой, а кто более древний). Но основываясь на двуххромосомные виды, можно сказать, так как существует вид Vibrio с Ala tRNA_GCA с Id=00049 и Ala tRNA_GCС с Id=00043; а также существуют виды Vibrio с Ala tRNA_GCA с Id=00047, то первоночально существовали именно Ala tRNA_GCA с Id=00047 и Ala tRNA_GCС с Id=00043. А они содержались в Shewanella, и поэтому его нужно признать наиболее древним организмом, и положить в основу систематики рассмотренных здесь организмов.
Тогда можно сделать вывод, что от Shewanella по одной линии произошли Yersinia. От Yersinia в свою очередь Photorhabdus, от которого и произошло все семейство Enterobacteriaceae. Но это только по одной линии. По другой же мы уже упоминали какие роды произошли от Shewanella.
Многовидовое происхождение сильно запутывает эволюционную картину, но с этим ничего не поделаешь — такова сложность видообразования, и нам нужно лишь их наиболее точно отразить в условиях, когда не все виды известны.
Автор: tac