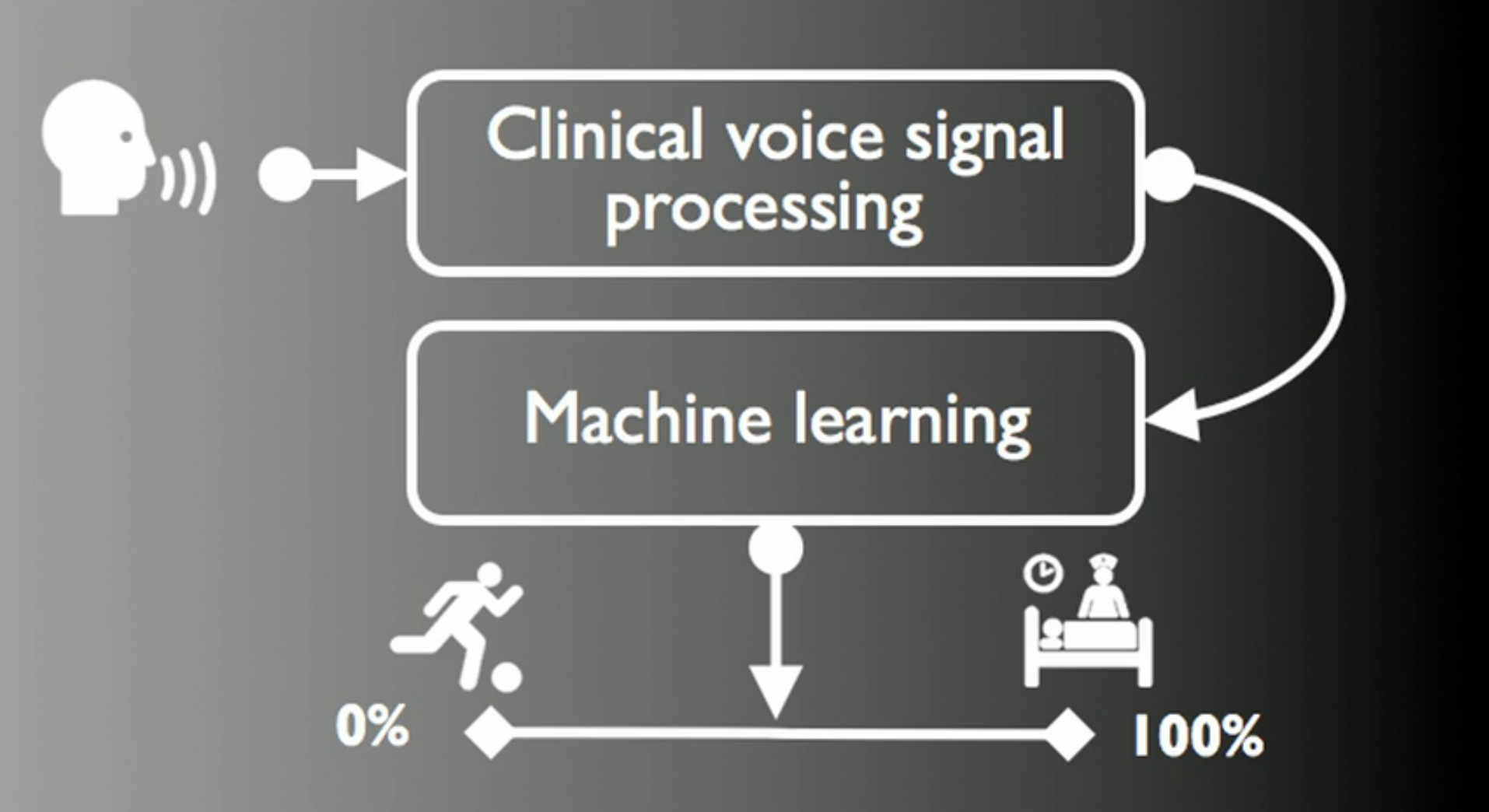
Из выступления Max Little (основателя PVI) на конференции TED в 2012 году.
Здравствуй, Хабрахабр. Данный цикл статей будет посвящен рассмотрению возможности и построению Open Source универсального анализатора нарушений речи.
В данной статье будет рассказано о проекте Parkinson Voice Initiative, посвященному ранней диагностике Болезни Паркинсона по голосу (успешность распознавания составляет 98,6± 2.1% за 30 секунд по телефонному разговору).
Будет произведено сравнение точности используемых в нем алгоритмов выбора особенностей (ВО) – Feature Selection Algorithm – LASSO, mRMR, RELIEF, LLBFS.
Битва между Random Forest (RF) и Supported Vector Machine (SVM) за звание лучшего анализатора в данного рода приложениях.
Начало
Читая статьи по синтезу и распознаванию речи, нашел упоминание о том, что при болезни изменяется голос. Проверив очевидность факта, что я не первый догадался использовать распознавание речи для диагностики болезней (первые клиницисты определили некоторые features — особенности еще в 40-х годах прошлого века, записывая на магнитофонную ленту, а потом вручную анализируя), пошел по ссылкам Гугла. Одна из первых указывала на проект PVI.

Читать полностью »