Один из наиболее частых вопросов, с которыми сталкиваются специалисты по обработке и анализу данных — «Какой язык программирования лучше всего использовать для решения задач, связанных с машинным обучением?» Ответ на этот вопрос всегда приводит к сложному выбору между R, Python и MATLAB. Вообще говоря, никто не может дать объективный ответ, какой же язык программирования лучше. Конечно, язык, который вы выбираете для машинного обучения, зависит от ограничений конкретной задачи и данных, предпочтений самого специалиста и тех методов машинного обучения, которые нужно будет применить. Согласно опросу о любимом инструменте пользователей Kaggle для решения задач анализа данных R предпочитают 543 из 1714.
Сейчас в CRAN доступен 8341 пакет. Кроме CRAN, есть и другие репозитории с большим количеством пакетов. Синтаксис для установки любого из них прост: install.packages(“Name_Of_R_Package”).
Вот несколько пакетов, без которых вы вряд ли обойдетесь, как специалист по анализу данных: dplyr, ggplot2, reshape2. Безусловно, это не полный список. В этой статье мы подробнее остановимся на пакетах, применяемых в машинном обучении.
5. randomForest: собрать много деревьев в лес
Алгоритм случайного леса — один из наиболее широко используемых алгоритмов машинного обучения. Пакет randomForest используется для создания большого количества деревьев решений, после чего каждое наблюдаемое значение помещается в дерево. Результат, содержащий наибольшее количество значений, считается финальным. Для применения алгоритма randomForest нужно убедиться, что все переменные являются либо числовыми, либо факторными. Фактор не может иметь более 32 уровней, если используется randomForest.
Как вы, возможно, знаете, алгоритм случайного леса берет случайные наборы переменных и значений и строит много деревьев. В конце концов эти деревья объединяются, и на основании лучшего решения определяется класс зависимой переменной.
Давайте возьмем для примера набор данных iris, чтобы построить случайный лес с помощью пакета randomForest.
Rf_fit<-randomForest(formula=Species~., data=iris)
Нужно выполнить строку кода по аналогии с остальными пакетами, и можно применять алгоритм случайного леса. Давайте посмотрим, как он работает.
print(Rf_fit)
Call:
randomForest(formula = Species ~ ., data = iris)
Type of random forest: classification
Number of trees: 500
No. of variables tried at each split: 2
OOB estimate of error rate: 4.67%
Confusion matrix:
setosa versicolor virginica class.error
setosa 50 0 0 0.00
versicolor 0 47 3 0.06
virginica 0 4 46 0.08
importance(Rf_fit)
MeanDecreaseGini
Sepal.Length 10.200682
Sepal.Width 2.673111
Petal.Length 43.116951
Petal.Width 43.246585
Возможно, потребуется варьировать разные управляющие параметры randomForest, например, количество переменных в каждом дереве, количество деревьев, которые нужно построить, и т.д. Обычно специалисты по анализу данных выполняют несколько итераций и выбирают наилучшую комбинацию.
6. nnet: все дело в скрытых уровнях
Это наиболее широко используемый и легкий для восприятия пакет для работы с нейронными сетями, но его ограничение — один уровень узлов. Однако, согласно некоторым исследованиям, больше и не требуется, поскольку они не только не добавляют модели производительности, но и увеличивают время вычисления и сложность модели.
Этот пакет не предоставляет никаких специальных методов для определения количества узлов на скрытом уровне. Поэтому когда специалисты по работе с большими данными применяют nnet, всегда предполагается, что нужно указать значение, лежащее между количеством входных и выходных узлов. Пакет nnet предоставляет реализацию алгоритма искусственных нейронных сетей (Artificial Neural Networks, ANNs), который создан исходя из знания о том, как работает человеческий , на основе входных и выходных сигналов. ANNs широко применяется для прогнозирования в авиации. В частности, нейронные сети дают лучшие результаты прогнозирования с применением функций nnet, чем стандартных методов прогнозирования, таких как экспоненциальное сглаживание, регрессия, т.д.
В R есть много пакетов для построения нейронных сетей, например, nnet, neuralnet, RSNNS. Давайте снова воспользуемся для примера набором данных iris (подозреваю, он вам уже надоел). Попробуем спрогнозировать Species с nnet и посмотреть, как это выглядит.
nnet_model <- nnet(Species~., data=iris, size = 10)

В выходной нейронной сети можно увидеть 10 скрытых слоев, т.к. мы установили size=10 при построении нейронной сети.
К сожалению, легкого пути построить полученную нейронную сеть нет, но на github есть множество специальных функций для этой цели. Например, для построения сети выше была использована эта.
7. e1071: векторы как опора для вашей модели
Это очень важный пакет языка R, в котором есть специальные функции для реализации наивного классификатора Байеса (условная вероятность), метода опорных векторов (Support Vector Machines, SVM), Фурье-преобразования, нечеткой кластеризации и т.д. Скажем, первая реализация SVM в R была именно в пакете e1071. Отлично подходит для случаев, когда, допустим, нужно определить, какова вероятность того, что человек, купивший iPhone 6S, также купит чехол для него.
Этот вид анализа основан на условной вероятности, поэтому специалисты по анализу данных используют пакет e1071, где есть функции, реализующие наивный классификатор Байеса.
Метод опорных векторов пригодится, если ваш набор данных не разделяется в исходной размерности, и вам нужно привести данные к более высокой размерности, чтобы воспользоваться методами классификации или регрессии. SVM использует кернфункции (для оптимизации математических операций) и максимизирует расстояние между двумя классами.
Синтаксис функций, реализующих SVM, аналогичен:
svm_model <- svm(Species ~Sepal.Length + Sepal.Width, data=iris)
Для визуализации SVM воспользуемся функцией plot() с соответствующими данными:
plot(svm_model, data = iris[,c(1,2,5)])
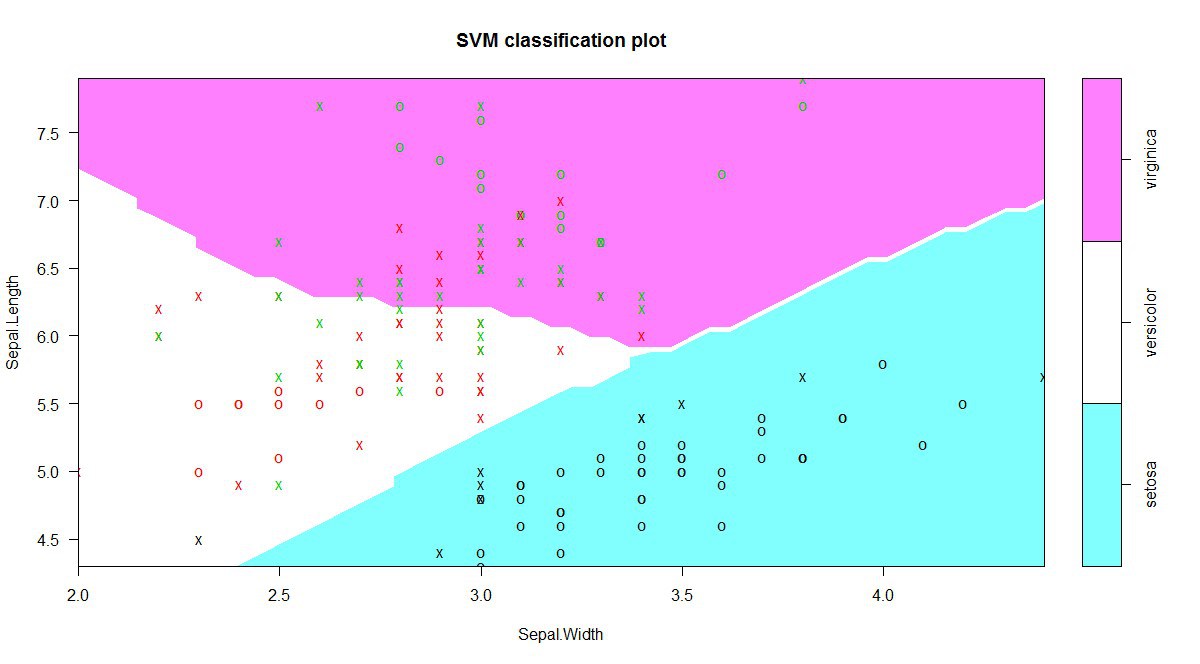
На графике выше четко видны границы, полученные после применения SVM к данным iris.
Есть множество параметров, которые вам, возможно, придется менять, чтобы получить наилучшую точность (kernel, cost, gamma, coefficients и др.). Для получения хорошей классификации с помощью SVM придется поэкспериментировать со многими из этих параметров: скажем, kernel может принимать разные значения — linear, Gaussian, Cosine.
8. kernLab: хорошо упакованное ядро
Kernlab использует преимущества объектной модели S4 в R, чтобы специалисты по анализу данных могли воспользоваться алгоритмами машинного обучения на основании ядер. В Kernlab есть реализации SVM, ядерного анализа, примитивов скалярного произведения, алгоритма ранжирования, гауссовых процессов и алгоритма спектральной кластеризации. Методы машинного обучения на основании ядер применяются, когда сложно решить задачи классификации, кластеризации и регрессии в пространстве результатов наблюдений.
Пакет Kernlab широко используется как реализация SVM, облегчающая задачу распознавания образов. Есть множество кернфункций, например, tanhdot (гиперболическая тангенсная кернфункция), polydot (полиномиальная кернфункция), laplacedot (кернфункция Лапласа) и другие, применяемые в распознавании образов.
Кернфункции чрезвычайно важны для SVM. Этот метод без них был бы невозможен.
SVM — не единственная техника, использующая ядра. Есть много других полезных и известных алгоритмов на основании ядер, таких как метод случайных вихрей (random vortex method, RVM), ядерный анализ главных компонент, уменьшение размерности, т.д. Пакет kernLab содержит порядка 20 таких алгоритмов. В kernLab есть свои предопределенные ядра, но пользователь может создавать и использовать свои собственные кернфункции.
Давайте инициализируем свою собственную радиальную базисную функцию среднеквадратическим значением 0,01.
Myrbf <- rbfdot(sigma = 0.01)
Myrbf
Gaussian Radial Basis kernel function.
Hyperparameter : sigma = 0.01
Можно узнать класс Myrbf, просто применив функцию class() к только что созданному объекту.
class(Myrbf)
[1] "rbfkernel"
attr(,"package")
[1] "kernlab"
Каждое ядро принимает на вход два вектора и возвращает их скалярное произведение. Давайте зададим два вектора и увидим их скалярное произведение.
x<-rnorm(10)
y<-rnorm(10)
Myrbf(x,y)
[,1]
[1,] 0.8443782
Мы создали две случайных нормально распределенных переменных, х и у, каждая с 10 значениями, и вычислили их скалярное произведение с помощью кернфункции Myrbf.
Давайте посмотрим на SVM с применением кернфункции Myrbf. Снова воспользуемся набором данных iris, чтобы понять, как работает SVM с применением kernLab.
Kernlab_svm <- ksvm(Species ~ Sepal.Length + Sepal.Width, data = iris, kernel = Myrbf, C=4)
Kernlab_svm
Support Vector Machine object of class "ksvm"
SV type: C-svc (classification)
parameter : cost C = 4
Gaussian Radial Basis kernel function.
Hyperparameter : sigma = 0.01
Number of Support Vectors : 103
Objective Function Value : -95.3715 -70.6262 -291.6249
Training error : 0.2
Давайте используем построенную только что SVM для прогнозирования:
predicted<-predict(Kernlab_svm,iris)
table(predicted = predicted, true = iris$Species)
true
predicted setosa versicolor virginica
setosa 49 0 0
versicolor 1 37 16
virginica 0 13 34
Заключение
Каждый пакет или функция в R имеет свои значения по умолчанию. Перед тем, как применять какой бы то ни было алгоритм, имеет смысл узнать, какие опции доступны. Значения по умолчанию дадут вам какой-то результат, но нет уверенности, что он будет наиболее оптимальным или точным.
В CRAN есть и другие пакеты для машинного обучения, например, igraph, glmnet, gbm, tree, CORElearn, mboost и др. Они применяются в различных сферах для построения наиболее эффективных моделей. Можно столкнуться с ситуациями, когда изменение одного параметра полностью поменяет вид выходных данных. Поэтому не стоит так уж полагаться на значения по умолчанию: изучите свои данные и требования, прежде чем применять какой-то алгоритм.
Автор: Инфопульс Украина